réservé à la recherche
Mouse Direct PCR Kit (For Genotyping)
Le kit Mouse Direct PCR permet une préparation rapide et une amplification PCR spécifiquement conçues pour le génotypage de souris.
De Selleck A été cité par 81 Publications
Comparaison de produits
Description
Le kit Mouse Direct PCR permet une préparation rapide et une amplification PCR spécifiquement conçues pour le génotypage de souris. Le Buffer L et la Protease Plus digèrent rapidement la souris
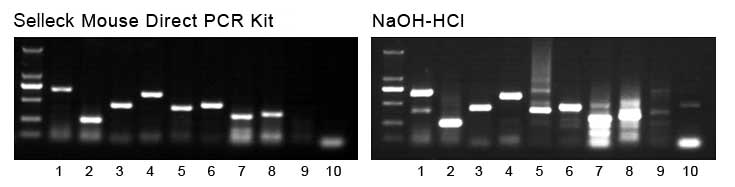
Les puits 1 à 10 représentent différents amorces PCR. Deux dentre elles servent de contrôles négatifs (puits 9 et 10) qui naffichent des bandes quen utilisant léchantillon de souris transgénique. Par conséquent, le kit Mouse Direct PCR assure une spécificité PCR plus élevée, ce qui en fait un produit fiable pour la recherche scientifique.
Analyse du résultat : Buffer L : Tampon de lyse. Protease Plus : Pour une digestion rapide et efficace du tissu de souris en seulement 15 minutes ! 2x M-PCR OPTITM Mix : Inclut la Taq ADN polymérase, les dNTP, le MgCl2 et le tampon de réaction optimisés et brevetés de Selleck.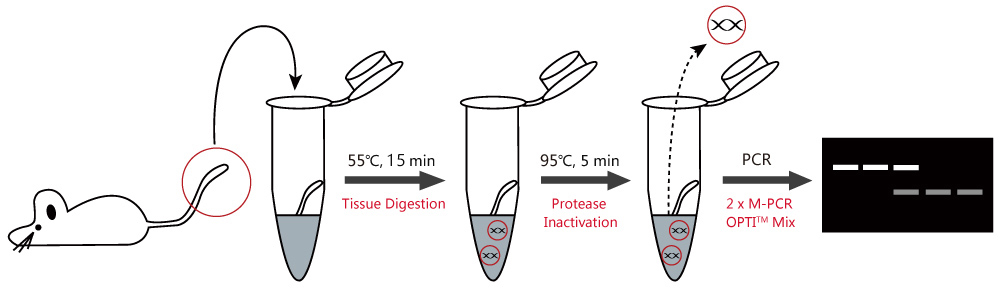
1. Comparaison entre différentes méthodes
Réactif
Prix ($)
Méthode de génotypage
NaOH-HCL
Proteinase K
Selleck Direct PCR Kit
Lysis Buffer
Sigma, 0.010
+
+
Buffer L
Proteinase K
Sigma P6556, 1U/reaction, 0.034
+
Protease Plus
Phenol Chloroform
Sigma P3803, 200μL/reaction, 0.116
+
+
Alcohols
Sigma I9030, 459836, 0.084
+
+
Taq
NEB M0273X, 1U/reaction, 0.122
+
+
2 x M-PCR OPTITM Mix
dNTP
Sigma GE28-4065-64, 0.25mM, 0.066
+
+
MgCL2, DNA Loading
Sigma, 0.010
+
+
Price ($) /Reaction
0.41
0.44
0.39
2. Comparaison des résultats PCR (souris de type sauvage)
3. Avantages
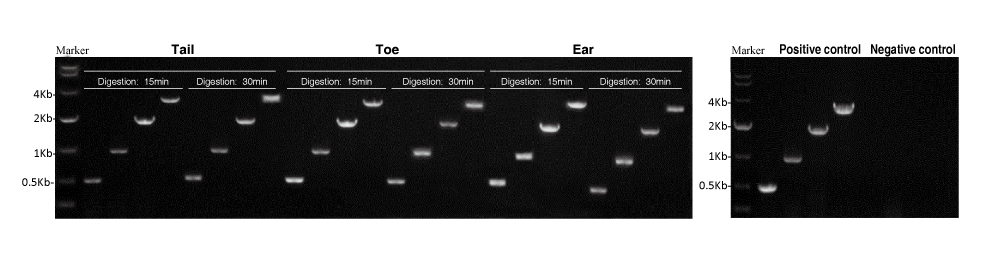
Le kit Mouse Direct PCR sapplique à la préparation déchantillons de queue, doreille et dorteil de souris. Aucune différence significative na été observée entre les groupes, qui digèrent à 55°C pendant 15 min ou 30 min. Le kit est également efficace pour lamplification de grands fragments (par exemple 3 Kb).
Remarque : Les amorces pour lamplification de fragments de 500 pb et 3 Kb sont conçues sur la base du gène GAPDH de la souris, tandis que les amorces pour les fragments de 1 Kb et 2 Kb sont conçues sur la base du gène Ppip5k2 de la souris.
4. Composants
Complete Kit
B40013 (200 rxns)
B40015 (500 rxns)
Buffer L
20 mL
25 mL x 2
Protease Plus
0.4 mL 1 mL
2 x M-PCR OPTITM Mix (Dye Plus)
1 mL x 2
1 mL x 5
User Guide
1
1
Stockage (à partir de la date de réception)
Le 2 x M-PCR OPTITM Mix et la Protease Plus doivent être conservés à -20°C pendant 2 ans. Si le PCR Mix doit être utilisé fréquemment, il peut être conservé à 4°C pendant 10 jours maximum.
Le Buffer L doit être conservé à 4°C.
Protocole
1. Protocole expérimental
1) Préparation de lADN génomique
Placez la queue, loreille ou lorteil de la souris dans un tube à centrifuger de 1,5 mL. Mélangez soigneusement 100 μL de Buffer L frais avec 2 μL de Protease Plus pour un seul échantillon dans un tube séparé. Ajoutez le mélange de protéase aux tubes de tissu de souris avec le tissu coupé immergé, puis incubez à 55°C pendant 15 minutes. Après le processus de digestion, incubez à 95°C pendant 5 minutes pour inactiver la protéase. Le lysat tissulaire peut maintenant être utilisé comme matrice PCR.
2) Génotypage PCR
Ajoutez de leau dd H2O, des amorces, la matrice et le 2 x M-PCR OPTITM Mix dans un tube PCR selon les concentrations recommandées. Mélangez rapidement au centrifugeuse et chargez dans lamplificateur PCR pour commencer lamplification. Il est recommandé de préparer la réaction PCR dans un bain de glace.
| Composants de la réaction PCR | 20 μL Volume de réaction (μL) | 50 μL Volume de réaction (μL) |
|---|---|---|
| ddH2O | 8 | 21 |
| Amorce avant (10 μM) | 0.5 | 1 |
| Amorce arrière (10 μM) | 0.5 | 1 |
| Matrice | 1 | 2 |
| 2 x M-PCR OPTI™ Mix | 10 | 25 |
| Étapes PCR | Température (°C) | Heure | Cycles |
|---|---|---|---|
| 1 | 94 | 5 min | 1 |
| 2 | 94 | 20 sec | 35 |
| 3 | 50-65 | 30 sec | |
| 4 | 72 | X min (2 kb /min) | |
| 5 | 72 | 5 min | 1 |
| 6 | 12 | -- | 1 |
Remarque : Avant la première utilisation
1. Tous les réactifs de ce kit ont été optimisés pour être utilisés ensemble et toute modification ou utilisation alternative est interdite.
2. Pour chaque étape, assurez-vous que chaque réactif du kit est bien mélangé avant utilisation.
3. Pendant létape de digestion tissulaire, agiter les tubes 1 à 2 fois sera utile pour libérer lADN génomique.
4. Pour la plupart des échantillons de tissus de souris, une incubation de 15 minutes à 55°C suffira pour lextraction de lADN génomique. Le tissu peut toujours sembler intact, mais lextraction a eu lieu.
5. LADN génomique de souris extrait doit être appliqué immédiatement avant létape damplification PCR. Un stockage inapproprié à long terme peut entraîner des amplifications PCR peu fiables.
Dépannage
Veuillez consulter les options de dépannage suivantes si vous rencontrez des difficultés techniques. Vous pouvez également contacter directement le support technique de Selleck.
| Problème | Cause(s) potentielle(s) | Suggestion(s) |
|---|---|---|
| Pas de produit damplification dans les échantillons de test ou de contrôle | La réaction damplification a été mal configurée | Optimisez la configuration correcte de la réaction |
| Un stockage inapproprié a entraîné une perte dactivité des réactifs PCR | Remplacez tous les composants par des réactifs frais | |
| Les amorces ne sont pas optimales et ne se sont pas hybridées | Redessiner les amorces | |
| Lamplification a fonctionné dans les échantillons de contrôle, mais pas dans les échantillons de test | La digestion était incomplète | Prolongez le temps de digestion jusquà 30 minutes à 55°C |
| Les échantillons ont été stockés trop longtemps et lADN génomique sest dégradé | Recueillir des échantillons frais de queue de souris pour lextraction dADN génomique | |
| La solution de lyse a été mélangée trop longtemps avec le mélange PCR | Les réactions PCR doivent être initiées dans les 5 heures suivant lajout de la matrice, et stockées à 4°C pendant ce temps | |
| La quantité de produit damplification nétait pas suffisante | Augmenter le nombre de cycles PCR à 35-40 pour obtenir plus de produit damplification | |
| Produit(s) damplification non spécifique(s) | La température dhybridation était trop basse | Augmenter la température dhybridation |
| Le nombre de cycles PCR était trop élevé | Diminuer le nombre de cycles à 30-35 | |
| La concentration damorce était trop élevée | Diminuer la concentration damorce | |
| La concentration de matrice était trop élevée | Diluer la matrice dans de leau purifiée H2O ou du tampon TE |
Support technique
Si vous avez dautres questions, veuillez laisser un message.
