Nur für Forschungszwecke
Mouse Direct PCR Kit (For Genotyping)
Das Mouse Direct PCR Kit ermöglicht eine schnelle Präparation und PCR-Amplifikation, die speziell für das Maus-Genotyping entwickelt wurde.
Sellecks Wurde zitiert von 81 Publikationen
Produktvergleich
Beschreibung
Das Mouse Direct PCR Kit ermöglicht eine schnelle Präparation und PCR-Amplifikation, die speziell für das Maus-Genotyping entwickelt wurde. Der Buffer L und die Protease Plus verdauen Maus
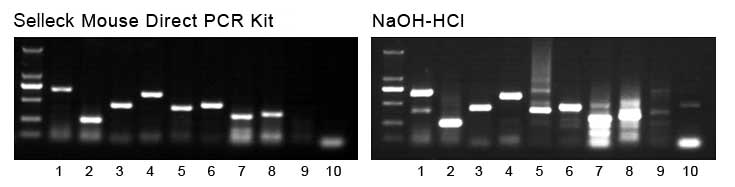
Spur 1-10 repräsentiert verschiedene PCR-Primer. Zwei davon dienen als Negativkontrollen (Spur 9 und 10), die nur bei Verwendung der transgenen Mausprobe Banden anzeigen. Daher gewährleistet das Mouse Direct PCR Kit eine höhere PCR-Spezifität, was es zu einem zuverlässigen Produkt für die wissenschaftliche Forschung macht.
Ergebnisanalyse: Buffer L: Lyse-Puffer. Protease Plus: Für den schnellen und effizienten Verdau von Mausgewebe in nur 15 Minuten! 2x M-PCR OPTITM Mix: Enthält Sellecks markenrechtlich geschützte und optimierte Taq-DNA-Polymerase, dNTPs, MgCl2 und Reaktionspuffer.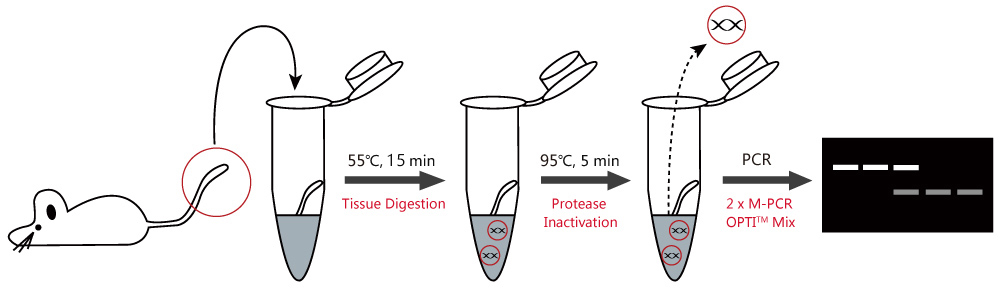
1. Vergleich zwischen verschiedenen Methoden
Reagenz
Preis ($)
Genotyping-Methode
NaOH-HCL
Proteinase K
Selleck Direct PCR Kit
Lysis Buffer
Sigma, 0.010
+
+
Buffer L
Proteinase K
Sigma P6556, 1U/reaction, 0.034
+
Protease Plus
Phenol Chloroform
Sigma P3803, 200μL/reaction, 0.116
+
+
Alcohols
Sigma I9030, 459836, 0.084
+
+
Taq
NEB M0273X, 1U/reaction, 0.122
+
+
2 x M-PCR OPTITM Mix
dNTP
Sigma GE28-4065-64, 0.25mM, 0.066
+
+
MgCL2, DNA Loading
Sigma, 0.010
+
+
Price ($) /Reaction
0.41
0.44
0.39
2. Vergleich der PCR-Ergebnisse (Wildtyp-Maus)
3. Vorteile
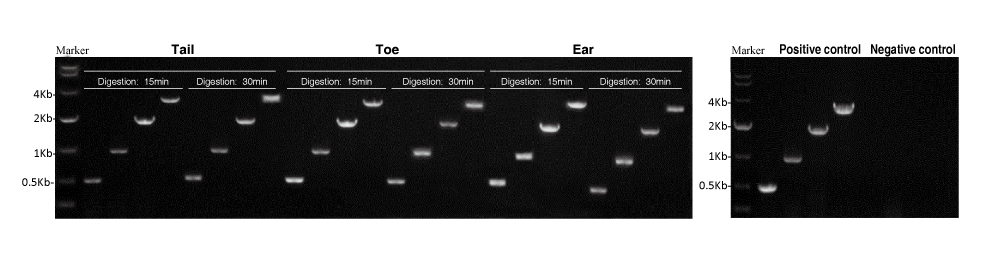
Das Mouse Direct PCR Kit ist für die Präparation von Proben aus Mausschwanz, -ohr und -zehe anwendbar. Es wurde kein signifikanter Unterschied zwischen den Gruppen beobachtet, die bei 55°C für 15 min oder 30 min verdaut werden. Das Kit ist auch für die Amplifikation großer Fragmente (z.B. 3 Kb) wirksam.
Hinweis: Die Primer für die Amplifikation von 500bp- und 3Kb-Fragmenten wurden basierend auf dem Maus-GAPDH-Gen entwickelt, während die Primer für 1Kb- und 2Kb-Fragmente basierend auf dem Maus-Ppip5k2-Gen entwickelt wurden.
4. Komponenten
Complete Kit
B40013 (200 rxns)
B40015 (500 rxns)
Buffer L
20 mL
25 mL x 2
Protease Plus
0.4 mL 1 mL
2 x M-PCR OPTITM Mix (Dye Plus)
1 mL x 2
1 mL x 5
User Guide
1
1
Lagerung (ab dem Datum des Erhalts)
2 x M-PCR OPTITM Mix und Protease Plus sollten bei -20°C für 2 Jahre gelagert werden. Wenn der PCR Mix häufig verwendet werden soll, kann er bis zu 10 Tage bei 4°C gelagert werden.
Buffer L sollte bei 4°C gelagert werden.
Protokoll
1. Experimentelles Protokoll
1) Genomische DNA-Präparation
Legen Sie Mausschwanz, -ohr oder -zehe in ein 1,5-ml-Zentrifugenröhrchen. Mischen Sie 100 μL frischen Buffer L gründlich mit 2 μL Protease Plus für eine einzelne Probe in einem separaten Röhrchen. Geben Sie die Proteasemischung zu den Mausgeweberöhrchen, wobei das Gewebe darin eingetaucht ist, und inkubieren Sie dann bei 55°C für 15 Minuten. Nach dem Verdauprozess inkubieren Sie bei 95°C für 5 Minuten, um die Protease zu inaktivieren. Das Gewebelysat kann nun als PCR-Template verwendet werden.
2) PCR-Genotyping
Geben Sie dd H2O, Primer, Template und 2 x M-PCR OPTITM Mix in ein PCR-Röhrchen gemäß den empfohlenen Konzentrationen. Zentrifugieren Sie die Mischung kurz und laden Sie sie in den PCR-Amplifikator, um die Amplifikation zu starten. Es wird empfohlen, die PCR-Reaktion im Eisbad vorzubereiten.
| PCR-Reaktionskomponenten | 20 μL Reaktionsvolumen (μL) | 50 μL Reaktionsvolumen (μL) |
|---|---|---|
| ddH2O | 8 | 21 |
| Forward Primer (10 μM) | 0.5 | 1 |
| Reverse Primer (10 μM) | 0.5 | 1 |
| Template | 1 | 2 |
| 2 x M-PCR OPTI™ Mix | 10 | 25 |
| PCR-Schritte | Temperatur (°C) | Zeit | Zyklen |
|---|---|---|---|
| 1 | 94 | 5 min | 1 |
| 2 | 94 | 20 sec | 35 |
| 3 | 50-65 | 30 sec | |
| 4 | 72 | X min (2 kb /min) | |
| 5 | 72 | 5 min | 1 |
| 6 | 12 | -- | 1 |
Hinweis: Vor dem ersten Gebrauch
1. Alle Reagenzien in diesem Kit wurden für die gemeinsame Verwendung optimiert, und jegliche Modifikationen oder alternative Verwendungen sind verboten.
2. Stellen Sie für jeden Schritt sicher, dass jedes Reagenz im Kit vor Gebrauch gut gemischt ist.
3. Während des Gewebeverdauungsschritts ist das 1-2-malige Schütteln der Röhrchen hilfreich, um die genomische DNA freizusetzen.
4. Für die meisten Mausgewebeproben reicht eine Inkubation von 15 Minuten bei 55°C für die genomische DNA-Extraktion aus. Das Gewebe kann immer noch intakt erscheinen, aber die Extraktion hat stattgefunden.
5. Die extrahierte genomische Maus-DNA sollte sofort vor dem PCR-Amplifikationsschritt verwendet werden. Eine unsachgemäße Langzeitlagerung kann zu unzuverlässigen PCR-Amplifikationen führen.
Fehlerbehebung
Bitte lesen Sie die folgenden Optionen zur Fehlerbehebung, wenn Sie auf technische Schwierigkeiten stoßen. Alternativ können Sie sich direkt an den technischen Support von Selleck wenden.
| Problem | Mögliche Ursache(n) | Vorschlag/Vorschläge |
|---|---|---|
| Kein Amplifikationsprodukt in Test- oder Kontrollproben | Die Amplifikationsreaktion wurde falsch eingerichtet | Optimieren Sie die richtige Reaktionseinrichtung |
| Unsachgemäße Lagerung hat zum Verlust der Aktivität von PCR-Reagenzien geführt | Ersetzen Sie alle Komponenten durch frische Reagenzien | |
| Primer sind nicht optimal und haben nicht annealiert | Primer neu gestalten | |
| Die Amplifikation funktionierte in den Kontrollproben, aber nicht in den Testproben | Der Verdau war unvollständig | Verlängern Sie die Verdauungszeit auf bis zu 30 Minuten bei 55°C |
| Proben wurden zu lange gelagert und die genomische DNA abgebaut | Sammeln Sie frische Mausschwanzproben für die genomische DNA-Extraktion | |
| Die Lyselösung wurde zu lange mit der PCR-Mischung gemischt | PCR-Reaktionen sollten innerhalb von 5 Stunden nach Zugabe des Templates gestartet und in der Zwischenzeit bei 4°C gelagert werden | |
| Die Menge des Amplifikationsprodukts war nicht ausreichend | Erhöhen Sie die Anzahl der PCR-Zyklen auf 35-40, um mehr Amplifikationsprodukt zu erhalten | |
| Unspezifisches Amplifikationsprodukt(e) | Die Annealing-Temperatur war zu niedrig | Erhöhen Sie die Annealing-Temperatur |
| Die Anzahl der PCR-Zyklen war zu hoch | Reduzieren Sie die Anzahl der Zyklen auf 30-35 | |
| Die Primerkonzentration war zu hoch | Primerkonzentration senken | |
| Die Template-Konzentration war zu hoch | Template in gereinigtem H2O oder TE-Puffer verdünnen |
Technischer Support
Wenn Sie weitere Anfragen haben, hinterlassen Sie bitte eine Nachricht.
