nur für Forschungszwecke
DBeQ p97 Inhibitor
Kat.-Nr.S7199
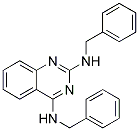
Chemische Struktur
Molekulargewicht: 340.42
Qualitätskontrolle
- In Nature Medicine für seine erstklassige Qualität zitiert
- COA
- NMR
- SDS
- Datenblatt
| Verwandte Ziele | Proteasome E1 Activating E3 Ligase DUB SUMO E2 conjugating |
|---|---|
| Weitere p97 Inhibitoren | NMS-873 CB-5339 ML240 NPD8733 |
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 340.42 | Formel | C22H20N4 |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 177355-84-9 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | JRF 12 | Smiles | C1=CC=C(C=C1)CNC2=NC(=NC3=CC=CC=C32)NCC4=CC=CC=C4 | ||
Löslichkeit
|
In vitro |
DMSO
: 68 mg/mL
(199.75 mM)
Ethanol : 5 mg/mL Water : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Merkmale |
Rapidly and potently induces activation of executioner caspases and cell death.
|
|---|---|
| Targets/IC50/Ki |
p97
1.5 μM
|
| In vitro |
DBeQ blockiert den Abbau von UbG76V-GFP, ODD-Luc und Luc-ODC mit IC50-Werten von 2,6 μM, 56 μM und 45 μM in HeLa-Zellen. Diese Verbindung ist mindestens 50-mal weniger potent gegenüber dem N-Ethylmaleimid-sensitiven Faktor (NSF) und dem 26S-Proteasom. Sie hemmt p97 kompetitiv in Bezug auf ATP mit einem Ki von 3,2 μM, was darauf hindeutet, dass sie an die aktive Stelle der D2-Domäne bindet. Diese Verbindung (10 μM) blockiert potent den Abbau von TCR
-GFP in HEK293-Zellen. Sie induziert CHOP innerhalb von 3 Stunden in einer konzentrationsabhängigen Weise, erhöht aber nicht den p21-Spiegel in HEK293-Zellen. Diese Verbindung (15 μM) induziert eine starke Akkumulation von LC3-II im Zellkern sowie in membranangereicherten und zytosolischen Fraktionen in Hela-Zellen. Sie wirkt, indem sie den autophagischen Abbau von LC3-II blockiert, anstatt die Autophagie in HeLa-Zellen zu induzieren. Diese Verbindung (10 μM) fördert schnell die Aktivierung der „Exekutor“-Kaspasen-3 und -7 in HeLa-Zellen. Sie aktiviert den intrinsischen Caspase-9-Apoptoseweg stärker als den extrinsischen Caspase-8-Weg, während STS beide Wege in ähnlichem Maße aktiviert. Diese Verbindung ist fünffach aktiver gegen multiple Myelom (RPMI8226)-Zellen als normale menschliche fetale Lungenfibroblasten (MRC5), wobei HeLa- und Hek293-Zellen intermediäre Empfindlichkeiten zeigen. Sie zeigt eine 20-fache Selektivität für die Stabilisierung von p97-abhängigen gegenüber unabhängigen UPS-Reporter-Substraten in HeLa-Zellen. Diese Chemikalie beeinträchtigt den Abbau von Substraten innerhalb der ERAD- und Autophagie-Wege. Sie (12 μM) hemmt die intrazelluläre Neutralisation dosisabhängig in HeLa-Zellen. Diese Verbindung (10 μM), die den Abbau von Virus und Antikörper im Fate-of-Capsid-Experiment vollständig hemmt, verhindert nicht den Abbau von IgG Fc. Sie (9 μM) reduziert den anfänglichen Neutralisationsgradienten als Funktion der Antikörperkonzentration. Diese Verbindung verringert sowohl die basale als auch die nährstoffstimulierte Phosphorylierung von MTOR-Zielen, ähnlich den Effekten von Rapamycin in U20S-Zellen.
|
| Kinase-Assay |
Manueller ATPase-Assay
|
|
Assay-Puffer [20 μL einer 2,5-fachen Konzentration, wobei 1× = 50 mM Tris (pH 7,4), 20 mM MgCl2, 1 mM EDTA und 0,5 mM Tris(2-carboxyethyl)phosphin (TCEP)] wird in jede Vertiefung einer 96-Well-Platte gegeben. Gereinigtes p97 (25 μL von 50 μM) wird in 975 μL 1× Assay-Puffer verdünnt, und 10 μL werden in jede Vertiefung gegeben. Diese Verbindung (10 μL) oder 5 % DMSO (10 μL) wird dann zu jeder Vertiefung hinzugefügt, und die Platte wird 10 Minuten lang bei Raumtemperatur inkubiert. Der ATPase-Assay wird durchgeführt, indem 10 μL von 500 μM ATP (pH 7,5) zu jeder Vertiefung hinzugefügt, 60 Minuten lang bei Raumtemperatur inkubiert und dann 50 μL Kinase Glo Plus-Reagenz hinzugefügt werden, gefolgt von einer abschließenden 10-minütigen Inkubation bei Raumtemperatur im Dunkeln. Die Lumineszenz wird auf einem Analyst AD gemessen. Diese Chemikalie wird in einer Reihe von Konzentrationen (0, 0,048, 0,24, 1,2, 6 und 30 μM) in dreifacher Ausführung getestet.
|
Literatur |
|
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
