nur für Forschungszwecke
YM201636 PIKfyve Inhibitor
Kat.-Nr.S1219
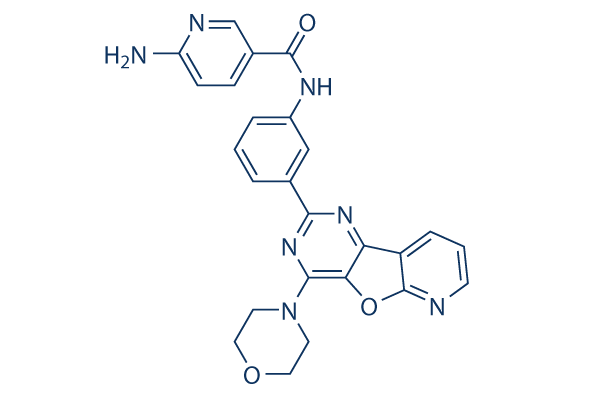
Chemische Struktur
Molekulargewicht: 467.48
Qualitätskontrolle
Zellkultur, Behandlung & Arbeitskonzentration
| Zelllinien | Assay-Typ | Konzentration | Inkubationszeit | Formulierung | Aktivitätsbeschreibung | PMID |
|---|---|---|---|---|---|---|
| SJ-GBM2 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for SJ-GBM2 cells | 29435139 | |||
| A673 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for A673 cells | 29435139 | |||
| SK-N-MC | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for SK-N-MC cells | 29435139 | |||
| BT-37 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for BT-37 cells | 29435139 | |||
| NB-EBc1 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for NB-EBc1 cells | 29435139 | |||
| U-2 OS | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for U-2 OS cells | 29435139 | |||
| Saos-2 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for Saos-2 cells | 29435139 | |||
| NB1643 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for NB1643 cells | 29435139 | |||
| LAN-5 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for LAN-5 cells | 29435139 | |||
| Rh18 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for Rh18 cells | 29435139 | |||
| OHS-50 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for OHS-50 cells | 29435139 | |||
| RD | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for RD cells | 29435139 | |||
| VERO-E6 | Toxicity assay | 48 hrs | Toxicity CC50 against VERO-E6 cells determined at 48 hours by high content imaging (same conditions as 2_LEY without exposure to 0.01 MOI SARS CoV-2 virus), CC50=0.01μM | ChEMBL | ||
| VERO-E6 | Function assay | 48 hrs | Determination of IC50 values for inhibition of SARS-CoV-2 induced cytotoxicity of VERO-E6 cells after 48 hours exposure to 0.01 MOI SARS CoV-2 virus by high content imaging, IC50=4.73μM | ChEMBL | ||
| Klicken Sie hier, um weitere experimentelle Daten zu Zelllinien anzuzeigen | ||||||
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 467.48 | Formel | C25H21N7O3 |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 371942-69-7 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | N/A | Smiles | C1COCCN1C2=NC(=NC3=C2OC4=C3C=CC=N4)C5=CC(=CC=C5)NC(=O)C6=CN=C(C=C6)N | ||
Löslichkeit
|
In vitro |
DMSO
: 12 mg/mL
(25.66 mM)
Water : Insoluble Ethanol : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Targets/IC50/Ki |
PIKfyve
(Cell-free assay) 33 nM
p110α
(Cell-free assay) 3.3 μM
|
|---|---|
| In vitro |
YM201636 hemmt potent PIKfyve von Säugetieren mit einer IC50 von 33 nM, jedoch nicht den Hefe-Ortholog Fab1 mit einer IC50 von >5 μM, was eine etwa 100-fache Selektivität für PtdIns3P p110α mit einer IC50 von 3 μM zeigt. Diese Verbindung (0,8 μM) verringert signifikant die Produktion von PtdIns(3,5)P2 um 80% in Serum-hungrigen NIH3T3-Zellen, gefolgt von Serumstimulation, ohne Auswirkung auf die Serum-stimulierte Proteinkinase B (PKB) Ser 473-Phosphorylierung. Sie beeinträchtigt reversibel den endosomalen Transport in NIH3T3-Zellen durch Blockierung von PIKfyve und PtdIns(3,5)P2-Produktion, was den Effekt der PIKfyve-Depletion mittels siRNA nachahmt. Diese Chemikalie (0,8 μM) reduziert auch signifikant das Knospen von Retroviren aus Zellen um 80%, anscheinend durch Interferenz mit dem endosomalen Sortierkomplex, der für den Transport (ESCRT)-Maschinerie erforderlich ist. In 3T3L1-Adipozyten hemmt sie die basale und Insulin-aktivierte 2-Desoxyglukose-Aufnahme mit einer IC50 von 54 nM, mit fast vollständiger Hemmung bei Dosen von nur 160 nM. Es wurde auch gezeigt, dass diese Verbindung (0,1 μM) die Insulin-abhängige Aktivierung der Klasse IA PI 3-Kinase vollständig blockiert. Obwohl nicht an der NPM-ALK-abhängigen Proliferation und Migration beteiligt, reduziert sie (0,4 μM) die invasiven Kapazitäten von NPM-ALK-exprimierenden Zellen und deren Fähigkeit, die extrazelluläre Matrix abzubauen, stark. Die Behandlung mit dieser Chemikalie blockiert das kontinuierliche Recycling der Junktionsproteine Claudin-1 und Claudin-2 in MDCK-Zellen, was zur intrazellulären Akkumulation und Verzögerung der epithelialen Barrierebildung führt.
|
Literatur |
|
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
