nur für Forschungszwecke
AS-605240 PI3K Inhibitor
Kat.-Nr.S1410
Chemische Struktur
Molekulargewicht: 257.27
Qualitätskontrolle
| Verwandte Ziele | Akt mTOR GSK-3 ATM/ATR DNA-PK AMPK PDPK1 PTEN PP2A PDK |
|---|---|
| Weitere PI3K Inhibitoren | GDC-0077 (Inavolisib) SAR405 Quercetin (Sophoretin) LY294002 XL147 analogue Tersolisib (STX-478) Buparlisib (BKM120) 740 Y-P (PDGFR 740Y-P) GO-203 TFA Eganelisib (IPI-549) |
Zellkultur, Behandlung & Arbeitskonzentration
| Zelllinien | Assay-Typ | Konzentration | Inkubationszeit | Formulierung | Aktivitätsbeschreibung | PMID |
|---|---|---|---|---|---|---|
| RAW264.7 cells | Function assay | Inhibition of MCP1-induced chemotaxis in mouse RAW264.7 cells, IC50=5.31 μM | ||||
| Klicken Sie hier, um weitere experimentelle Daten zu Zelllinien anzuzeigen | ||||||
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 257.27 | Formel | C12H7N3O2S |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 648450-29-7 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | N/A | Smiles | C1=CC2=NC=CN=C2C=C1C=C3C(=O)NC(=O)S3 | ||
Löslichkeit
|
In vitro |
DMSO
: 1 mg/mL
(3.88 mM)
Water : Insoluble Ethanol : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Merkmale |
The most potent member of a new class of PI3Kγ-selective inhibitors.
|
|---|---|
| Targets/IC50/Ki |
PI3Kγ
(Cell-free assay) 8 nM
PI3Kα
(Cell-free assay) 60 nM
PI3Kβ
(Cell-free assay) 270 nM
PI3Kδ
(Cell-free assay) 300 nM
|
| In vitro |
AS-605240 ist ein ATP-kompetitiver PI3Kγ-Inhibitor mit Ki-Werten von 7,8 nM. Diese Verbindung ist isoformselektiv, da sie auch PI3Kα, β und δ mit IC50-Werten von 60, 270 bzw. 300 nM hemmt. Sie hemmt die C5a-vermittelte PKB-Phosphorylierung mit einer IC50 von 90 nM. In Knochenmark-abgeleiteten Monozyten (BMDMs) blockiert diese Chemikalie (1 μM) die MCP-1- oder CSF-1-induzierte PKB-Phosphorylierung. An SC-CA1-Synapsen in Mäusen eliminiert diese Verbindung (100 nM) die NMDAR-LTD, ohne mGluR-LTD, Depotenzierung und LTP zu beeinflussen.
|
| Kinase-Assay |
In-vitro-PI3K-Lipidkinase-Assay
|
|
(1) Für PI3Kγ: humane PI3Kγ (100 ng) wird bei RT mit Kinasepuffer (10 mM MgCl2, 1 mM β-Glycerophosphat, 1 mM DTT, 0,1 mM Na3VO4, 0,1% Na-Cholat und 15 μM ATP/100 nCi γ[33P]ATP, Endkonzentrationen) und Lipidvesikeln, die 18 μM PtdIns und 250 μM PtdSer (Endkonzentrationen) enthalten, in Gegenwart von AS-605240 oder DMSO inkubiert. Die Kinase-Reaktion wird durch Zugabe von 250 μg Neomycin-beschichteter Scintillation Proximity Assay (SPA)-Beads gestoppt. (2) Für PI3Kα, β und δ: variable Mengen ATP werden mit den verschiedenen gereinigten PI3K-Isoformen und sättigenden Konzentrationen von PtdIns inkubiert. Folglich werden IC50-Bestimmungen mit PI3Kα, β und δ zur Bewertung der Inhibitorselektivität wie folgt durchgeführt: 60 ng PI3Kα werden bei RT mit Kinasepuffer, wie für PI3Kγ beschrieben (jedoch mit 89 μM ATP/300 nCi γ[33P]ATP und ohne Na-Cholat) und Lipidvesikeln, die 212 μM PtdIns und 58 μM PtdSer enthalten, inkubiert. 100 ng PI3Kβ werden bei RT mit Kinasepuffer (enthaltend 70 μM ATP/300 nCi γ[33P]ATP, 4 mM MgCl2 und ohne Na-Cholat) und Lipidvesikeln, die 225 μM PtdIns und 45 μM PtdSer enthalten, inkubiert. 90 ng PI3Kδ werden mit Kinasepuffer (enthaltend 65 μM ATP/300 nCi γ[33P]ATP, 1 mM MgCl2 und ohne Na-Cholat) und Lipidvesikeln, die 100 μM PtdIns und 170 μM PtdSer enthalten, inkubiert. Die Reaktionen werden nach 2 Stunden gestoppt.
|
|
| In vivo |
In einem RANTES-induzierten Mausmodell der Peritonitis reduziert AS-605240 die Neutrophilen-Chemotaxis mit einer ED50 von 9,1 mg/kg. Bei einer αCII-induzierten Arthritis schützt diese Verbindung (50 mg/kg) vor αCII-IA-Symptomen. In einem Mausmodell der Kollagen-induzierten Arthritis unterdrückt diese Verbindung (50 mg/kg) auch Gelenkentzündungen und -schäden. In einem Adipositas-induzierten Diabetesmodell (ob/ob-Mäuse) senkt diese Verbindung (10 mg/kg) den Blutzuckerspiegel, verbessert signifikant sowohl die Insulinsensitivität als auch die Glukosetoleranz, ohne das Körpergewicht zu beeinflussen. Diese Chemikalie (30 mg/kg) zeigt tiefgreifendere Effekte mit einer geringeren Gewichtszunahme. Darüber hinaus reduziert diese Verbindung die Häufigkeit von ATMs und die zirkulierenden MCP-1-Spiegel.
|
Literatur |
|
Anwendungen
| Methoden | Biomarker | Bilder | PMID |
|---|---|---|---|
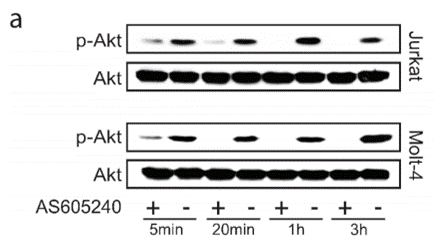
| Western blot | p-AKT / AKT |
|
25869207 |
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
