nur für Forschungszwecke
Taselisib (GDC 0032) PI3K Inhibitor
Kat.-Nr.S7103
Chemische Struktur
Molekulargewicht: 460.53
Qualitätskontrolle
| Verwandte Ziele | Akt mTOR GSK-3 ATM/ATR DNA-PK AMPK PDPK1 PTEN PP2A PDK |
|---|---|
| Weitere PI3K Inhibitoren | GDC-0077 (Inavolisib) SAR405 Quercetin (Sophoretin) LY294002 XL147 analogue Tersolisib (STX-478) Buparlisib (BKM120) 740 Y-P (PDGFR 740Y-P) GO-203 TFA Eganelisib (IPI-549) |
Zellkultur, Behandlung & Arbeitskonzentration
| Zelllinien | Assay-Typ | Konzentration | Inkubationszeit | Formulierung | Aktivitätsbeschreibung | PMID |
|---|---|---|---|---|---|---|
| human MOLM16 cells | Proliferation assay | 72 h | Antiproliferative activity against human MOLM16 cells after 72 hrs by Cell Titer-Blue assay | |||
| Klicken Sie hier, um weitere experimentelle Daten zu Zelllinien anzuzeigen | ||||||
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 460.53 | Formel | C24H28N8O2 |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 1282512-48-4 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | RG7604 | Smiles | CC1=NN(C(=N1)C2=CN3CCOC4=C(C3=N2)C=CC(=C4)C5=CN(N=C5)C(C)(C)C(=O)N)C(C)C | ||
Löslichkeit
|
In vitro |
DMSO
: 70 mg/mL
(151.99 mM)
Ethanol : 7 mg/mL Water : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Merkmale |
A beta isoform-sparing PI3K inhibitor.
|
|---|---|
| Targets/IC50/Ki |
PI3Kδ
(Cell-free assay) 0.12 nM(Ki)
PI3Kα
(Cell-free assay) 0.29 nM(Ki)
PI3Kγ
(Cell-free assay) 0.97 nM(Ki)
PI3Kβ
(Cell-free assay) 9.1 nM(Ki)
C2β
(Cell-free assay) 292 nM
hVps34
(Cell-free assay) 374 nM
|
| In vitro |
GDC-0032 ist ein oral bioverfügbarer, potenter und selektiver Inhibitor der Klasse I PI3Kα-, δ- und γ-Isoformen, mit 30-fach geringerer Hemmung der PI3K β-Isoform relativ zur PI3Kα-Isoform. Präklinische Daten zeigen, dass GDC-0032 eine erhöhte Aktivität gegen PI3Kα-Isoform (PIK3CA)-mutierte und HER2-amplifizierte Krebszelllinien aufweist. GDC-0032 hemmt die Proliferation von MCF7-neo/HER2-Zellen mit einer IC50 von 2,5 nM. |
| Kinase-Assay |
Charakterisierung der biochemischen und zellulären Aktivität in vitro
|
|
Die enzymatische Aktivität der Klasse-I-PI3K-Isoformen wird unter Verwendung eines Fluoreszenzpolarisationsassays gemessen, der die Bildung des Produkts 3,4,5-Inositoltriphosphat-Moleküls überwacht, während es mit fluoreszenzmarkiertem PIP3 um die Bindung an das GRP-1-Pleckstrin-Homologie-Domänenprotein konkurriert. Eine Zunahme des Phosphatidylinositid-3-Phosphat-Produkts führt zu einer Abnahme des Fluoreszenzpolarisationssignals, da der markierte Fluorophor vom GRP-1-Proteinbindungsort verdrängt wird. Klasse-I-PI3K-Isoformen werden als heterodimere rekombinante Proteine exprimiert und gereinigt. Tetramethylrhodamin-markiertes PIP3 (TAMRA-PIP3), di-C8-PIP2 und PIP3-Detektionsreagenzien werden von Echelon Biosciences gekauft. PI3Kα wird unter Ausgangsbedingungen in Gegenwart von 10 mM Tris (pH 7,5), 25 μM ATP, 9,75 μM PIP2, 5% Glycerin, 4 mM MgCl2, 50 mM NaCl, 0,05% (v/v) Chaps, 1 mM Dithiothreitol und 2% (v/v) DMSO bei 60 ng/mL getestet. Nach 30-minütigem Assay bei 25 °C werden die Reaktionen mit einer Endkonzentration von 9 mM EDTA, 4,5 nM TAMRA-PIP3 und 4,2 μg/mL GRP-1-Detektorprotein beendet, bevor die Fluoreszenzpolarisation auf einem Envision-Plattenleser abgelesen wird. IC50-Werte werden aus der Anpassung der Dosis-Wirkungs-Kurven an eine 4-Parameter-Gleichung berechnet. Jeder gemeldete Wert ist ein Durchschnitt aus drei Experimenten, und alle haben eine Standardabweichung von weniger als einem geometrischen Mittelwert.
|
|
| In vivo |
Die Pharmakokinetik von GDC-0032 ist annähernd dosisproportional und zeitunabhängig mit einer mittleren t1/2 von 40 Stunden. Die Kombination von GDC-0032 erhöht die Aktivität, was zu Tumorregressionen und einer Verzögerung des Tumorwachstums führt (91% Tumorwachstumsinhibition (TGI)). Darüber hinaus erhöht die Kombination von GDC-0032 die Wirksamkeit in vivo (102% TGI für GDC-0032). |
Literatur |
Anwendungen
| Methoden | Biomarker | Bilder | PMID |
|---|---|---|---|
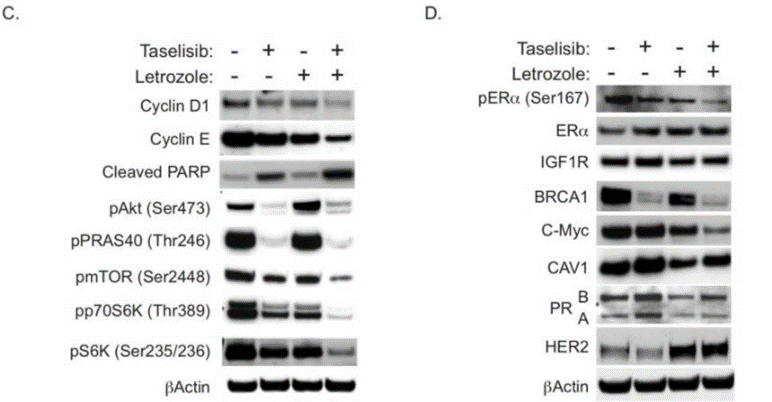
| Western blot | Cyclin D1 / Cyclin E / Cleaved PARP / p-AKT / pPRAS40 / p-mTOR / pp70S6K / BRCA1 / c-Myc |
|
27382432 |
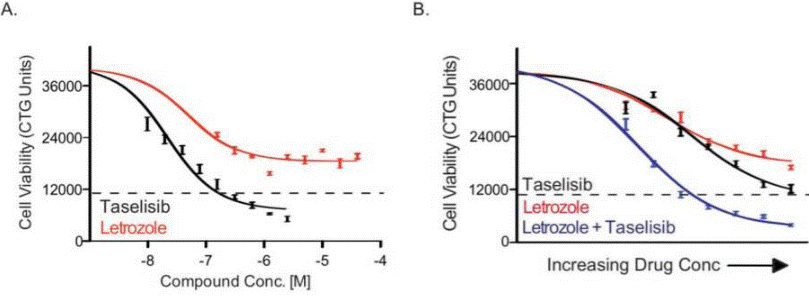
| Growth inhibition assay | Cell viability |
|
27382432 |
Klinische Studieninformationen
(Daten von https://clinicaltrials.gov, aktualisiert am 2024-05-22)
| NCT-Nummer | Rekrutierung | Erkrankungen | Sponsor/Kooperationspartner | Startdatum | Phasen |
|---|---|---|---|---|---|
| NCT02785913 | Completed | Recurrent Squamous Cell Lung Carcinoma|Stage IV Squamous Cell Lung Carcinoma |
SWOG Cancer Research Network|National Cancer Institute (NCI) |
November 2014 | Phase 2 |
| NCT01967966 | Completed | Healthy Volunteer |
Genentech Inc. |
November 2013 | Phase 1 |
| NCT01814709 | Completed | Healthy Volunteer |
Genentech Inc. |
April 2013 | Phase 1 |
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
