nur für Forschungszwecke
RI-1 RAD51 Inhibitor
Kat.-Nr.S8077
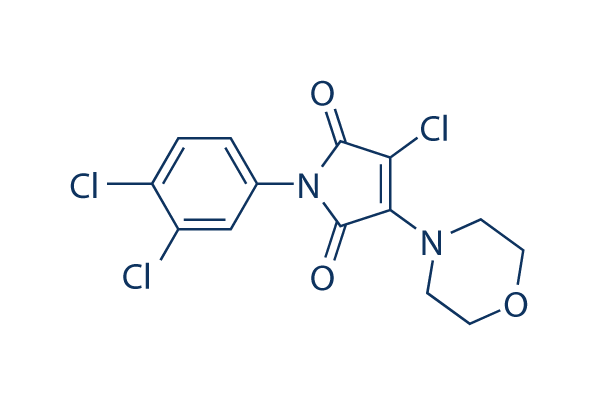
Chemische Struktur
Molekulargewicht: 361.61
Qualitätskontrolle
- In Nature Medicine für seine erstklassige Qualität zitiert
- COA
- NMR
- HPLC
- SDS
- Datenblatt
| Verwandte Ziele | HDAC PARP ATM/ATR DNA-PK WRN DNA/RNA Synthesis Topoisomerase PPAR Sirtuin Casein Kinase |
|---|---|
| Weitere RAD51 Inhibitoren | RS-1 Emzadirib RAD51-IN-17 |
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 361.61 | Formel | C14H11Cl3N2O3 |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 415713-60-9 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | RAD51 inhibitor 1 | Smiles | C1COCCN1C2=C(C(=O)N(C2=O)C3=CC(=C(C=C3)Cl)Cl)Cl | ||
Löslichkeit
|
In vitro |
DMSO
: 50 mg/mL
(138.27 mM)
Water : Insoluble Ethanol : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Merkmale |
A selective recombinant RAD51 protein inhibitor discovered in 2012. Valuable tool for mechanistic studies of DNA repair and potential for use in many cancers.
|
|---|---|
| Targets/IC50/Ki |
RAD51
<30 μM
|
| In vitro |
RI-1 sensibilisiert Zellen für DNA-Schäden, indem es HsRAD51 direkt und spezifisch stört und die Fähigkeit von RAD51, Filamente auf ssDNA zu bilden, hemmt. Darüber hinaus erzeugt diese Verbindung allein eine Toxizität als Einzelwirkstoff in allen drei Krebszelllinien (HeLa, MCF-7 und U2OS) mit LD50-Werten im Bereich von 20–40 µM. Es verringert das Wiederverbinden von γ-H2AX-Foci in G2-Phasen-Zellen und führt 6 Stunden nach Bestrahlung zu einem höheren Grad an nicht reparierten DSBs.
|
| Kinase-Assay |
DNA-Bindungsassays
|
|
Alle Reaktionen werden in schwarzen, nicht bindenden Polystyrol-384-Well-Platten mit Reaktionsvolumina von 30–100 μL durchgeführt. Gereinigte DNA-Strangaustauschproteine und chemische Verbindungen werden 5 Minuten lang bei Raumtemperatur vorinkubiert; anschließend werden sie weitere 30 Minuten bei 37 °C mit 100 nM ssDNA-Substrat inkubiert, das aus einem 45-mer Poly-dT besteht, das am 5'-Terminus mit Alexa 488 markiert ist (synthetisiert und gereinigt von Integrated DNA Technologies). Die Reaktionen werden in 20 mM HEPES pH 7,5, 10 mM MgCl2, 0,25 μM BSA, 2 % Glycerin, 30 mM NaCl, 4 % DMSO und 2 mM ATP durchgeführt. Einige Bedingungen enthielten DTT oder TCEP (Tris(2-carboxyethyl)phosphin), wie angegeben. Die DNA-Bindung wird als Funktion der Fluoreszenzpolarisation (FP) mit einem Safire2-Plattenlesegerät gemessen, wobei die folgenden Einstellungen verwendet werden: Anregung 470±5nm, Emission 530±5nm, 10 Messungen/Well, Z-Höhe und G-Faktor automatisch aus Kontrollwells kalibriert. Die angezeigten Fehlerbalken stellen die Standardabweichung dar. Für Experimente, die eine Titration von Proteinkonzentrationen beinhalten, werden die Daten an eine Gleichung angepasst, die die kooperative Natur berücksichtigt, mit der Rekombinaseproteine DNA binden. Für Experimente, die eine Titration dieser Verbindung beinhalten, werden Proteinkonzentrationen so gewählt, dass eine Sättigung des FP-Signals von ∼80 % in Abwesenheit dieser Chemikalie erreicht wird.
|
Literatur |
|
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
