nur für Forschungszwecke
TTNPB (Arotinoid acid) RAR Agonist
Kat.-Nr.S4627
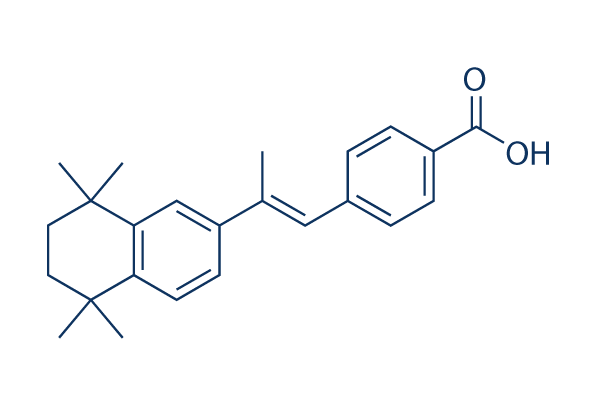
Chemische Struktur
Molekulargewicht: 348.48
Qualitätskontrolle
| Verwandte Ziele | Dehydrogenase HSP Transferase P450 (e.g. CYP17) PDE phosphatase PPAR Vitamin Carbohydrate Metabolism Mitochondrial Metabolism |
|---|---|
| Weitere Retinoid Receptor Inhibitoren | Tamibarotene AM580 Fenretinide UVI 3003 SR 11237 AR7 BMS493 All trans-Retinal MSU-42011 Palovarotene |
Zellkultur, Behandlung & Arbeitskonzentration
| Zelllinien | Assay-Typ | Konzentration | Inkubationszeit | Formulierung | Aktivitätsbeschreibung | PMID |
|---|---|---|---|---|---|---|
| HEK293 cells | Function assay | Agonist activity at human RARalpha expressed in HEK293 cells by luciferase reporter gene assay, EC50=0.18 nM | ||||
| CV-1 cells | Function assay | Binding affinity against retinoic Acid gamma receptors co-transfected into CV-1 cells, EC50=0.002 Μm | ||||
| HL60 cells | Cytotoxicity assay | In vitro cytotoxicity against HL60 cells, IC50=46 μM | ||||
| HeLa cells | Function assay | Agonist activity at human RARalpha expressed in human HeLa cells assessed as relative luminescence units at >=10 uM by luciferase assay relative to control | ||||
| Klicken Sie hier, um weitere experimentelle Daten zu Zelllinien anzuzeigen | ||||||
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 348.48 | Formel | C24H28O2 |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 71441-28-6 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | Arotinoid Acid, Ro 13-7410, AGN-191183 | Smiles | CC(=CC1=CC=C(C=C1)C(=O)O)C2=CC3=C(C=C2)C(CCC3(C)C)(C)C | ||
Löslichkeit
|
In vitro |
DMSO
: 15 mg/mL
(43.04 mM)
Water : Insoluble Ethanol : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Targets/IC50/Ki |
RARβ
(cell-free assay) 4.5 nM
RARα
(cell-free assay) 5.1 nM
RARγ
(cell-free assay) 9.3 nM
|
|---|---|
| In vitro |
TTNPB bindet mit hoher Affinität an nukleäre Retinsäurerezeptoren und hemmt die Bindung von [3H]tRA mit einer IC50 von 3,8 nM, 4,0 nM bzw. 4,5 nM für mRARα, β und γ. Diese Verbindung erhöht die transkriptionelle Aktivierung von Maus-RARs in JEG-3-Zellen nach 72 Stunden unter Verwendung von konditioniertem Medium mit einer EC50 von 2,0 nM, 1,1 nM bzw. 0,8 nM für mRARα, β und γ. Sie hemmt das Wachstum normaler menschlicher Brustepithelzellen (HMECs) und Östrogenrezeptor-positiver (ER-positiver) Brustkrebszellen, indem sie eine G1-Zellzyklusblockade induziert. Diese Chemikalie verursacht eine konzentrationsabhängige Abnahme der Differenzierung von ES-D3-Zellen.
|
| Kinase-Assay |
Bindungstests
|
|
Bindungstests werden wie zuvor beschrieben durchgeführt (Allenby et al., 1993, 1994). Kurz gesagt, markierte und unmarkierte Retinoide werden in Ethanol zu Nukleosol- oder zytosolischen Fraktionen gegeben, so dass die Gesamtmenge des zugesetzten Ethanols in allen Röhrchen konstant ist und 2 % des Inkubationsvolumens nicht überschreitet. Die Rezeptorpräparate werden bei 4 °C für 4–6 Stunden mit Retinoiden inkubiert. Sephadex PD-10 Entsalzungssäulen werden verwendet, um gebundenes Radioligand von freiem Radioligand zu trennen, nachdem das Gleichgewicht erreicht ist. Für kompetitive Bindungstests werden variierende Konzentrationen von unmarkiertem kompetitivem Ligand mit dem entsprechenden Nukleosol oder Zytosol in Gegenwart einer festen Konzentration von [3H]tRA (sp. Akt. 49,3 Ci/mmol) oder [3H]9-cis RA (sp. Akt. 24,0 Ci/mmol) inkubiert. Die Endkonzentrationen von [3H]tRA und [3H]9-cis RA für nukleäre Rezeptor-Bindungstests betragen 5 nM. Die Endkonzentrationen von [3H]tRA für CRABP-Bindungstests betragen 30 nM. Die IC50s werden wie oben beschrieben berechnet (DeLean et al., 1978). Für die Sättigungskinetik werden zunehmende Konzentrationen von radiomarkiertem Liganden ([3H]tRA sp. Akt. 49,3 Ci/mmol, [3H]this compound sp. Akt. 5,5 Ci/mmol) zu dem Nukleosol des entsprechenden Rezeptorsubtyps in Anwesenheit (unspezifische Bindung) oder Abwesenheit (Gesamtbindung) eines 100-fachen molaren Überschusses des entsprechenden unmarkierten Retinoids hinzugefügt. Spezifische Bindung ist definiert als die Gesamtbindung minus unspezifische Bindung. Sättigungskinetiken werden wie zuvor beschrieben berechnet (Scatchard, 1949; Grippo und Gudas, 1987; Levin et al., 1992).
|
|
| In vivo |
TTNPB (0,25 mg/kg) verursacht Wachstumshemmung in beiden MXT-HS- und MXT-HI-Modellen durch Induktion von Zellapoptose.
|
Literatur |
|
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
