nur für Forschungszwecke
D 4476 Casein Kinase Inhibitor
Kat.-Nr.S7642
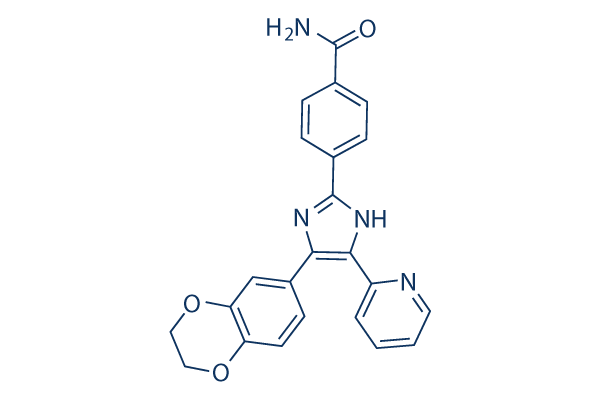
Chemische Struktur
Molekulargewicht: 398.41
Qualitätskontrolle
| Verwandte Ziele | Dehydrogenase HSP Transferase P450 (e.g. CYP17) PDE phosphatase PPAR Vitamin Carbohydrate Metabolism Mitochondrial Metabolism |
|---|---|
| Weitere Casein Kinase Inhibitoren | Silmitasertib (CX-4945) Silmitasertib (CX-4945) sodium salt TBB IC261 PF-670462 Ellagic Acid hydrate TTP 22 Longdaysin PF 4800567 (E/Z)-GO289 |
Zellkultur, Behandlung & Arbeitskonzentration
| Zelllinien | Assay-Typ | Konzentration | Inkubationszeit | Formulierung | Aktivitätsbeschreibung | PMID |
|---|---|---|---|---|---|---|
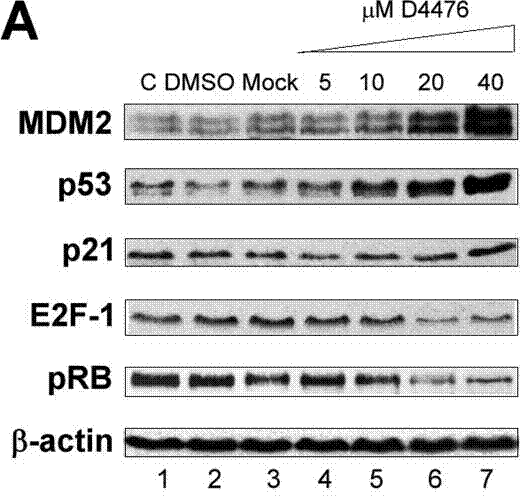
| A375 cells | Function assay | 10-80 μM | 48 h | Inhibition of CK1-MDM2 complex in human A375 cells assessed as increase in p21 protein level at 10 to 80 uM after 48 hrs by immunoblotting analysis | ||
| Klicken Sie hier, um weitere experimentelle Daten zu Zelllinien anzuzeigen | ||||||
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 398.41 | Formel | C23H18N4O3 |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 301836-43-1 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | N/A | Smiles | C1COC2=C(O1)C=CC(=C2)C3=C(NC(=N3)C4=CC=C(C=C4)C(=O)N)C5=CC=CC=N5 | ||
Löslichkeit
|
In vitro |
DMSO
: 79 mg/mL
(198.28 mM)
Water : Insoluble Ethanol : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Targets/IC50/Ki |
CK1 from Schizosaccharomyces pombe
200 nM
CK1δ
300 nM
ALK5
500 nM
|
|---|---|
| In vitro |
D4476 (10 μM) hemmt CK1 und ALK5 effektiv mit Aktivitäten von etwa 7 % bzw. 22 % im Vergleich zur Kontrollgruppe. In H4IIE-Hepatomzellen hemmt D4476 spezifisch die Phosphorylierung des endogenen Forkhead-Box-Transkriptionsfaktors O1a (FOXO1a) an Ser322 und Ser325, ohne die Phosphorylierung anderer Stellen zu beeinflussen. D4476 induziert Zytotoxizität in den Linien ANBL6, INA6 und RPMI8226; die Linien MM1S und U266 sind weniger empfindlich; und die Linie OPM1 ist vollständig resistent. Hohe Konzentrationen (50 μM) von D4476 induzierten Toxizität in allen Multiplen Myelom (MM)-Linien. In MM-Zellen erhöht D4476 die Proteinspiegel von TP53, P27 und FADD sowie die Zellzyklusprogression und die Induktion der Apoptose. Die Behandlung von Leukämie-Stammzellen (LSCs) mit D4476 zeigt eine hochselektive Abtötung von LSCs gegenüber normalen HSPCs.
|
| Kinase-Assay |
CK1 kinase assay
|
|
Alle Proteinkinase-Assays (25 μL) werden bei Raumtemperatur (21℃) durchgeführt. Die Assays werden 40 min lang mit einer Biomek 2000 Laboratory Automation Workstation in einem 96-Well-Format durchgeführt. Die Konzentrationen von Magnesiumacetat und [γ-33P]ATP (800 cpm/pmol) in den Assays betragen 10 mM bzw. 0,1 mM. Die Assays werden mit MgATP gestartet und durch Zugabe von 5 μL 0,5 M Orthophosphorsäure gestoppt. Aliquots werden dann auf P30-Filtermatten getupft, viermal in 75 mM Phosphorsäure gewaschen, um ATP zu entfernen, einmal in Methanol, dann getrocknet und auf Radioaktivität gezählt. CK1 δ (5-20 m-Einheiten), verdünnt in 20 mM Hepes, pH 7,5, 0,15 M NaCl, 0,1 mM EGTA, 0,1 % (v/v) Triton X-100, 5 mM Dithiothreit, 50 % (v/v) Glycerin, wird gegen das Peptid RRKDLHDDEEDEAMSITA in einer Inkubation getestet, die 20 mM Hepes, pH 7,5, 0,15 M NaCl, 0,1 mM EDTA, 5 mM DTT, 0,1 % (v/v) Triton X-100 und 0,5 mM Substratpeptid enthält.
|
Literatur |
|
Anwendungen
| Methoden | Biomarker | Bilder | PMID |
|---|---|---|---|
| Western blot | MDM2 / p53 / p21 / E2F-1 / pRB |
|
19759023 |
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
