nur für Forschungszwecke
P5091 DUB Inhibitor
Kat.-Nr.S7132
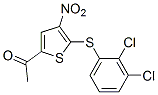
Chemische Struktur
Molekulargewicht: 348.22
Qualitätskontrolle
| Verwandte Ziele | Proteasome E1 Activating E3 Ligase p97 SUMO E2 conjugating |
|---|---|
| Weitere DUB Inhibitoren | PR-619 IU1 P22077 b-AP15 ML323 LDN-57444 VLX1570 TCID EOAI3402143 ML364 |
Zellkultur, Behandlung & Arbeitskonzentration
| Zelllinien | Assay-Typ | Konzentration | Inkubationszeit | Formulierung | Aktivitätsbeschreibung | PMID |
|---|---|---|---|---|---|---|
| Sf9 cells | Function assay | Inhibition of recombinant USP7 expressed in Sf9 cells by Ub-CHOP reporter assay, EC50=4.2 μM | 24900381 | |||
| human HCT116 cells | Cytotoxic assay | 72 h | Cytotoxicity against human HCT116 cells after 72 hrs, EC50=11 μM | 24900381 | ||
| multiple myeloma cells | Function assay | Inhibition of USP7 in multiple myeloma cells (unknown origin) by immunohistochemistry, EC50 = 4.2 μM. | 25364867 | |||
| Sf9 | Function assay | 30 mins | Inhibition of full-length recombinant USP7 (unknown origin) expressed in Sf9 cells using Ub-EKL as substrate preincubated for 30 mins followed by substrate addition by fluorescence based assay, IC50 = 4.2 μM. | 28768102 | ||
| Klicken Sie hier, um weitere experimentelle Daten zu Zelllinien anzuzeigen | ||||||
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 348.22 | Formel | C12H7Cl2NO3S2 |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 882257-11-6 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | P005091 | Smiles | CC(=O)C1=CC(=C(S1)SC2=C(C(=CC=C2)Cl)Cl)[N+](=O)[O-] | ||
Löslichkeit
|
In vitro |
DMSO
: 28 mg/mL
(80.4 mM)
Water : Insoluble Ethanol : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Targets/IC50/Ki |
USP7
(Cell-free assay) 4.2 μM(EC50)
USP47
(Cell-free assay) 4.3 μM
|
|---|---|
| In vitro |
P5091 ist ein trisubstituiertes Thiophen mit Dichlorphenylthio-, Nitro- und Acetyl-Substituenten, die die Anti-USP7-Aktivität vermitteln. Diese Verbindung zeigt eine potente, spezifische und selektive Deubiquitinierungsaktivität gegen USP7. Im Gegensatz dazu hemmt sie keine anderen DUBs oder andere getestete Familien von Cysteinproteasen (EC50 > 100 mM). Diese Chemikalie hemmt die Markierung von USP7 mit HA-UbVME in einer konzentrationsabhängigen Weise. Die USP7-vermittelte Spaltung von hochmolekularen Polyubiquitinketten wird durch diese Verbindung dosisabhängig gehemmt. Darüber hinaus hemmt sie die USP7-, aber nicht die USP2- oder USP8-vermittelte Spaltung von Poly-K48-verknüpften Ubiquitinketten. Die USP7-Hemmung durch diese Verbindung induziert die Polyubiquitinierung von HDM2 und beschleunigt den Abbau von HDM2. Sie hemmt die USP7-Deubiquitinierungsaktivität, ohne die Proteasomaktivität in MM-Zellen zu blockieren. Diese Chemikalie induziert einen dosisabhängigen Rückgang der Lebensfähigkeit verschiedener MM-Zelllinien. Sie überwindet das durch Knochenmark-Stromazellen induzierte Wachstum von MM-Zellen. Diese Verbindung verringerte HDM2 und HDMX sowie erhöhte p53- und p21-Spiegel. Insgesamt wird die durch diese Verbindung induzierte Zytotoxizität teilweise über die HDM2-p21-Signalachse vermittelt, und obwohl p53 als Reaktion auf diese chemische Behandlung hochreguliert wird, ist die zytotoxische Aktivität davon nicht p53-abhängig. |
| Kinase-Assay |
Ubiquitin Protease-Assays
|
|
Rekombinante Enzyme in 20 mM Tris-HCl (pH 8,0), 2 mM CaCl2 und 2 mM β-Mercaptoethanol werden mit Dosisbereichen von P5091 für 30 min in einer 96-Well-Platte inkubiert, bevor Ub-PLA2 und NBD C6-HPC oder Ub-EKL und EKL-Substrat hinzugefügt werden. Die Freisetzung eines fluoreszierenden Produkts innerhalb des linearen Bereichs des Assays wird mit einem Perkin Elmer Envision Fluoreszenzplattenleser überwacht. Vehikel (2 % [v/v] DMSO) und 10 mM N-Ethylmaleimid (NEM) sind als Kontrollen enthalten.
|
|
| In vivo |
In Tiermodellstudien für Tumore wird P5091 gut vertragen, hemmt das Tumorwachstum und verlängert das Überleben. Die Kombination dieser Verbindung mit dem HDAC-Inhibitor SAHA löst eine synergistische Anti-MM-Aktivität aus. |
Literatur |
|
Anwendungen
| Methoden | Biomarker | Bilder | PMID |
|---|---|---|---|
| Western blot | USP7 / p53 / p21 HAUSP / N-Myc |
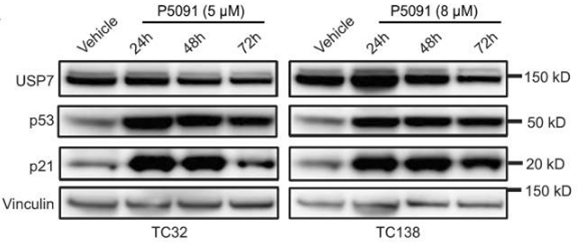
|
30045945 |
| Immunofluorescence | PTEN |
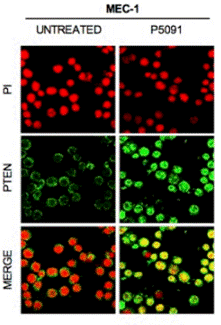
|
28418900 |
| Growth inhibition assay | Cell viability |
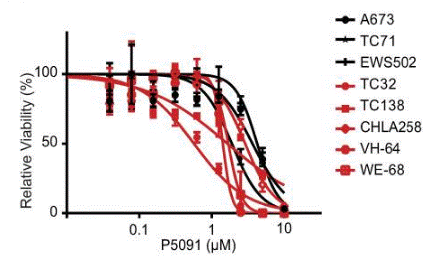
|
30045945 |
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
