nur für Forschungszwecke
T0901317 Liver X Receptor Agonist
Kat.-Nr.S7076

Chemische Struktur
Molekulargewicht: 481.33
Qualitätskontrolle
| Verwandte Ziele | Dehydrogenase HSP Transferase P450 (e.g. CYP17) PDE phosphatase PPAR Vitamin Carbohydrate Metabolism Mitochondrial Metabolism |
|---|---|
| Weitere Liver X Receptor Inhibitoren | GW3965 Hydrochloride LXR-623 (WAY-252623) GSK2033 SR9243 Abequolixron (RGX-104) AZ876 GSK3987 Desmosterol |
Zellkultur, Behandlung & Arbeitskonzentration
| Zelllinien | Assay-Typ | Konzentration | Inkubationszeit | Formulierung | Aktivitätsbeschreibung | PMID |
|---|---|---|---|---|---|---|
| THP1 cells | Function assay | Stimulation of [3H]cholesterol efflux in human THP1 foam cells loaded with ac-LDL, EC50=3 nM | ||||
| COS7 cells | Function assay | 16 h | Agonist activity at human LXRbeta receptor transfected in COS7 cells after 16 hrs by reporter transactivation assay, EC50=11 nM | |||
| CHO cells | Function assay | Agonist activity at human LXR beta receptor expressed in CHO cells by reporter assay, EC50=16 nM | ||||
| HEK293 cells | Function assay | Agonist activity at human LXRbeta in HEK293 cells assessed as Gal4 transactivation, EC50=19 nM | ||||
| mouse J774 cells | Function assay | Agonist activity at LXRbeta in mouse J774 cells assessed as upregulation of ABCA1 mRNA expression, EC50=0.027 μM | ||||
| mouse J774A1 cells | Function assay | 18 h | Induction of ABCA1 mRNA expression in mouse J774A1 cells after 18 hrs by RT-PCR, EC50=0,034 μM | |||
| HuH7 cells | Function assay | Upregulation of SREB1c mRNA level in human HuH7 cells, EC50=0.036 μM | ||||
| HepG2 cells | Function assay | Effect on SREBP1c gene expression in human HepG2 cells, EC50=0.061 μM | ||||
| SH-SY5Y cells | Function assay | 24 h | Agonist activity at human LXRbeta expressed in human SH-SY5Y cells co-transfected with Gal4-LBD after 24 hrs by luciferase reporter gene assay, EC50=0.075 μM | |||
| CV1 cells | Function assay | Transactivation of LXRbeta (unknown origin) expressed in CV1 cells by luciferase reporter gene assay, EC50=0.14 μM | ||||
| human H4 cells | Function assay | Effect on gamma-secretase activity in human H4 cells expressing APP695 assessed as increase in Amyloid beta-42 formation LPECL assay, EC50=3.6 μM | ||||
| Klicken Sie hier, um weitere experimentelle Daten zu Zelllinien anzuzeigen | ||||||
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 481.33 | Formel | C17H12F9NO3S |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 293754-55-9 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | N/A | Smiles | C1=CC=C(C=C1)S(=O)(=O)N(CC(F)(F)F)C2=CC=C(C=C2)C(C(F)(F)F)(C(F)(F)F)O | ||
Löslichkeit
|
In vitro |
DMSO
: 96 mg/mL
(199.44 mM)
Ethanol : 96 mg/mL Water : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Targets/IC50/Ki |
LXR
(Cell reporter assay) 50 nM(EC50)
FXR
5 μM(EC50)
|
|---|---|
| In vitro |
T0901317 wirkt ber LXR und induziert in Verbindung mit seinem RXR-Heterodimerisierungspartner die Expression des ABCA1-Reverse-Cholesterintransporters. Diese Verbindung reguliert die Expression des ABCA1-Gens hoch, das mit der Cholesterin-Efflux-Regulation und dem HDL-Metabolismus assoziiert ist. Es zeigt einen EC50-Wert von ~ 5 M f die Aktivierung von Gallens ure-Farnesoid-X-Rezeptoren (FXRs), 10-fach potenter als der nat liche FXR-Ligand Chenodeoxychols ure. Diese Chemikalie ist auch ein hochaffiner Ligand f den Xenobiotika-Rezeptor Pregnan-X-Rezeptor (PXR). Es bindet und aktiviert PXR mit der gleichen nanomolaren Potenz, mit der es die LXR-Aktivit t stimuliert. Diese Verbindung induziert die Expression nicht nur von LXR-Zielgenen, sondern auch von PXR-Zielgenen in Zellen und Tieren, einschlie lich des Scavenger-Rezeptors CD36. Es verringert die Amyloid- -Produktion in prim ren Neuronen in vitro. Es wurde festgestellt, dass diese Chemikalie direkt mit ROR und ROR mit hoher Affinit t bindet (Ki = 132 bzw. 51 nM), was zur Modulation der F higkeit des Rezeptors f hrt, mit transkriptionellen Kofaktorproteinen zu interagieren. Es unterdr ckt die ROR / -abh ngige Transaktivierung von ROR-responsiven Reportergenen und reduziert in HepG2-Zellen die Rekrutierung des Steroidrezeptor-Koaktivators-2 durch ROR an einem endogenen ROR-Zielgen (G6Pase). |
| In vivo |
Die T0901317-Behandlung von 11 Wochen alten APP23-M usen f 6 Tage zeigt einen signifikanten Anstieg der ABCA1-Expression und eine Abnahme des Verh ltnisses von l slichen APP (sAPP) - zu sAPP -Spaltprodukten. Am wichtigsten ist, dass diese Verbindung eine statistisch signifikante Reduzierung der Spiegel von l slichem A 40 und von A 42 im Gehirn dieser M u se bewirkt. |
Literatur |
|
Anwendungen
| Methoden | Biomarker | Bilder | PMID |
|---|---|---|---|
| Western blot | LXR-β / LXR-α EGFR / p-PI3K p55 / p-PI3K p85 / PI3K p85 / p-PDK1 / PDK1 |
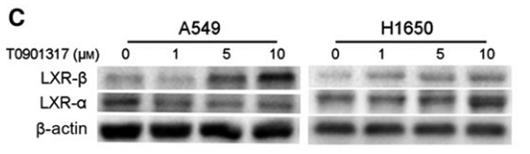
|
28097086 |
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
