nur für Forschungszwecke
JIB-04 Histone Demethylase Inhibitor
Kat.-Nr.S7281
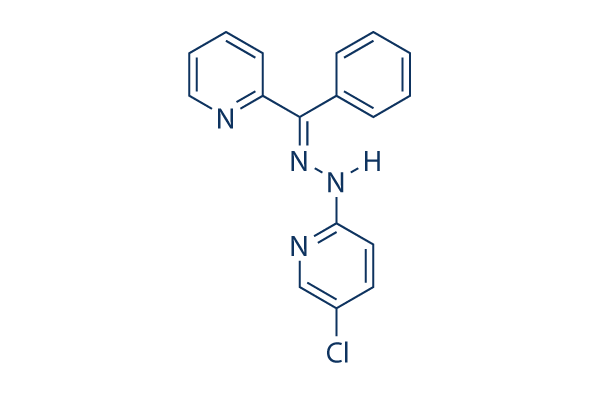
Chemische Struktur
Molekulargewicht: 308.76
Qualitätskontrolle
| Verwandte Ziele | HDAC JAK BET Histone Methyltransferase PKC PARP HIF PRMT EZH2 AMPK |
|---|---|
| Weitere Histone Demethylase Inhibitoren | GSK-J4 Hydrochloride SP2509 GSK-LSD1 2HCl Ladademstat (ORY-1001) Dihydrochloride CPI-455 HCl OG-L002 IOX1 GSK J1 ML324 CPI-455 |
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 308.76 | Formel | C17H13ClN4 |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 199596-05-9 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | NSC 693627 | Smiles | C1=CC=C(C=C1)C(=NNC2=NC=C(C=C2)Cl)C3=CC=CC=N3 | ||
Löslichkeit
|
In vitro |
DMSO
: 62 mg/mL
(200.8 mM)
Water : Insoluble Ethanol : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Targets/IC50/Ki |
JARID1A
(Cell-free assay) 230 nM
JMJD2D
(Cell-free assay) 290 nM
JMJD2E
(Cell-free assay) 340 nM
JMJD2B
(Cell-free assay) 435 nM
JMJD2A
(Cell-free assay) 445 nM
JMJD3
(Cell-free assay) 855 nM
|
|---|---|
| In vitro |
JIB-04 induziert transkriptionelle Veränderungen auf krebsselektive Weise, einschließlich der Herunterregulierung von proliferativen Genen und der Hochregulierung von antiproliferativen/pro-apoptotischen Genen. Diese Verbindung blockiert das Wachstum von Lungen- und Prostatakrebszellen mit einer IC50 von nur 10 nM, während sie weniger antiproliferative Aktivitäten auf HBECs und PrSCs/PrECs hervorruft.
|
| Kinase-Assay |
Jumonji Demethylase-/Prolylhydroxylase-/LSD1-Aktivitätsassays
|
|
Aktives JMJD2E aa 1-350 wird aus E. coli gereinigt und in vitro in Gegenwart von α-Ketoglutarat, 5-10 μM Eisen, Ascorbinsäure und einem Histonpeptidsubstrat in einer gekoppelten Reaktion mit Formaldehyddehydrogenase, ergänzt mit NAD+, verwendet, um die NADH-Produktion zu quantifizieren oder mit dem Epigentek-Kit P-3081. Für Histon-Demethylierungsreaktionen, die mittels Western-Analyse quantifiziert werden, wird ein His-getaggtes hJMJ2D aa 1-350 Expressionskonstrukt, das freundlicherweise von Dr. Y. Shi und J. Whetstine zur Verfügung gestellt wurde, exprimiert und aus E. coli nach den Anweisungen des Qiagen Ni-NTA Agarose-Handbuchs und dem Protokoll von Whestine et al. 54 gereinigt. Kurz gesagt, das Protein wird in 50 mM TrisHCl pH 7,8 + 0,3 M NaCl + 10 % Glycerin + 200 mM Imidazol eluiert und gegen 20 mM TrisHCl pH 7,4, 0,15 M NaCl, 0,2 mM PMSF, 0,5 mM DTT, 8 % Glycerin dialysiert. Das Enzym wird aliquotiert, schockgefroren und bei -80 °C gelagert. Für Aktivitätsassays mittels Western Blot werden ~1,5 μg Enzym mit 0,3 μg H3K9me3-Substrat (Active Motif #31213) in 10 μM (NH4)2Fe(SO4)2, 1 mM α-Ketoglutarat und 2 mM Natrium-L-Ascorbat in 50 mM Hepes pH 7,9 in Gegenwart von Vehikel oder Medikament kombiniert und 30 Minuten bis 2 Stunden bei 37 °C inkubiert. SDS-Probenpuffer wird zu den Reaktionen gegeben, und nach dem Kochen werden die Proben auf NuPage 4-12 % Bis-Tris-Gelen aufgetragen, auf Nitrozellulose übertragen und mit Upstate #07-523 geblottet, um H3K9me3 nachzuweisen. Zum Nachweis des H3-Gesamtsignals verwenden wir Active Motif #39763 (primär) und IRDye 680 konjugiertes Esel-Anti-Kaninchen-IgG (sekundär, LI-COR # 926-32223) und bildeten die Blots in einem Odyssey Infrarot-Bildgebungssystem ab, das freundlicherweise von Dr. M. Cobb zur Verfügung gestellt wurde. Für in vitro IC50-Bestimmungen und Konkurrenzstudien werden typischerweise 100-200 ng gereinigtes Protein mit Vehikel, JIB-04 oder Analoga, wie in den Legenden der Abbildungen angegeben, inkubiert und die Aktivität mittels ELISA gemessen (Epigentek-Kit P-3081 für H3K9me3-Demethylierung, P-3083 für H3K4me3-Demethylierung und P-3085 für H3K27me3-Demethylierung) in Reaktionen, die 50 mM Hepes pH 7,5, 0,01 % Tween 20, 120 nM (NH4)2Fe(SO4)2, 1 mM α-Ketoglutarat, 2 mM Natrium-L-Ascorbat und 50 ng Peptidsubstrat enthalten. Die endgültigen Enzymkonzentrationen in den Reaktionen waren wie folgt: 206 nM JMJD2A, 12 nM JMJD2B, 60 nM JMJD2C, 90 nM JMJD2D E.coli, 30 nM JMJD2D Sf9, 30 nM JMJD2E, 30 nM Jarid1a, 35 nM JMJD3. Hintergrundmessungen werden durch hitzeinaktivierte Enzyme, 0,5-1 mM 2,4 PDCA oder Reaktionen ohne 2-OG erhalten. hJMJD2A (aa1-350), gereinigt in E.coli, ist ein freundliches Geschenk von Dr. Jose Rizo-Rey und wird mit 400 ng/Reaktion aufgrund seiner intrinsisch geringen Aktivität getestet. GraphPad Prism Software wird für IC50-Berechnungen und Kurvenanpassung verwendet. E.coli JMJD2D, von uns gereinigt, und Sf9 JMJD2D von BPS lieferten nicht unterscheidbare Ergebnisse. Beachten Sie, dass für Substratkonkurrenzassays, um im linearen Bereich des Assays zu bleiben und die Bindungskapazität der ELISA-Platte nicht zu sättigen, Reaktionen mit > 0,75 μM H3K9me3 unbiotinyliertes Substrat enthalten oder im Detektionsschritt verdünnt und die Signale pro Verdünnungsfaktor angepasst werden. Für die direkte Quantifizierung der H3K9me3-Demethylaseaktivität in Zelllysaten wurden behandelte Zellen (plattiert mit 2 Millionen/10cm-Schale) oder Tumorhomogenate in PBS beschallt (3x 4 Sek.) und gleiche Proteinmengen wurden mit einem Histon-H3K9me3-Substrat in einem Reaktionspuffer mit Kofaktoren für 2 Stunden bei 37 °C inkubiert, bevor eine spezifische Immun-Detektion des H3K9me2-Produkts mit Epigentek-Kit P-3081-Reagenzien erfolgte. 500 ng E.coli gereinigtes PHD2-Protein in 40 mM Tris pH 7,4, 100 mM NaCl, 20 % Glycerin, 5 mM β-Mercaptoethanol, 10 mM Maltose werden verwendet, um Aktivität im linearen Bereich zu erhalten. Biotinylierte Peptide, die vom HIF-1 ODD stammen (Biotin-Acp-DLDLEALAPYIPADDDFQL oder Biotin-Acp-DLDLEALAP(OH)YIPADDDFQL als hydroxylierte Kontrolle), werden auf Neutravidin-beschichteten 96-Well-Platten immobilisiert. Das Enzym wird in den beschichteten Wells in Reaktionspuffer (20 mM Tris-Cl pH 7,5, 5 mM KCl, 1,5 mM MgCl2, 2 mM DTT, 0,12 μM Eisen(II)sulfat, 0,5 mM 2-Oxoglutarat und 1 mM Ascorbat) für 45 Minuten bei Raumtemperatur in Gegenwart der angegebenen Medikamente inkubiert. Das kompetitive Analogon von α-Ketoglutarat, DMOG, wird als positive Kontrolle für die Hemmung verwendet. Die Peptidhydroxylierung wird unter Verwendung eines polyklonalen Kaninchenantikörpers nachgewiesen, der gegen ein hydroxyliertes HIF-Peptidepitop (Kaninchen-Anti-Hydroxyprolin 4817, Eigenproduktion) erzeugt wurde, gefolgt von der Zugabe eines Ziegen-Anti-Kaninchen-HRP-konjugierten Sekundärantikörpers. Die Lumineszenz wird in einem EnVision-Plattenlesegerät gemessen. Die Aktivität des rekombinanten LSD1-Proteins wird mit dem Epigentek-Kit P-3075 gemäß dem Herstellerprotokoll mit dem proprietären Inhibitor gemessen.
|
|
| In vivo |
In zwei separaten Xenograft-Mausmodellen (H358 oder A549) verringert JIB-04 das Tumorwachstum, senkt die Jumonji histone demethylase-Aktivität in Tumoren und verlängert das Krebsüberleben.
|
Literatur |
Anwendungen
| Methoden | Biomarker | Bilder | PMID |
|---|---|---|---|
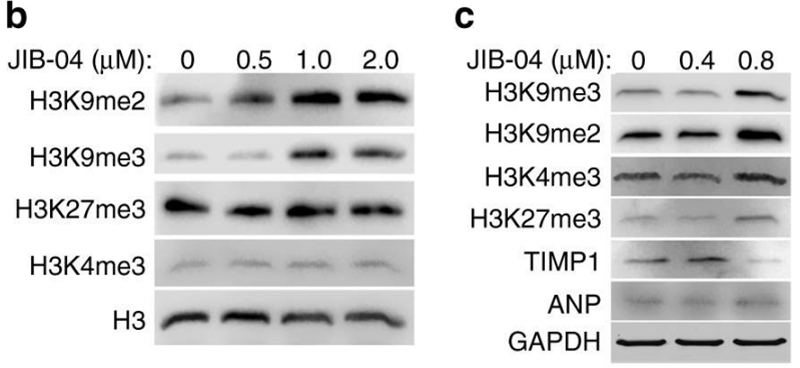
| Western blot | H3K9me2 / H3K9me3 / H3K27me3 / H3K4me3 / TIMP1 / ANP αCD133 |
|
30531796 |
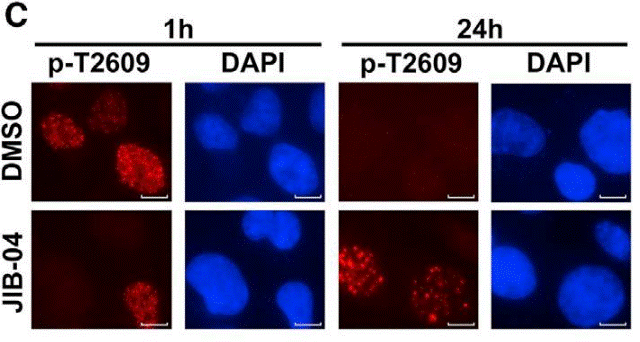
| Immunofluorescence | DNA-PKcs |
|
30355483 |
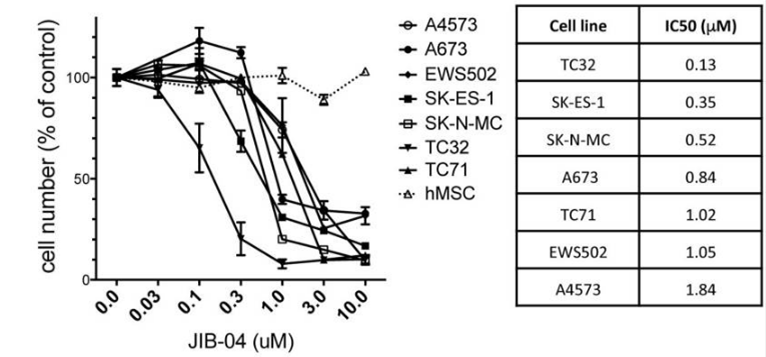
| Growth inhibition assay | Cell viability |
|
30237855 |
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
Häufig gestellte Fragen
Frage 1:
Do you have any suggestion for in vivo study please? (via injection)
Antwort:
For IP or sub cutaneous injection, this compound is soluble in 4% DMSO+30% PEG 300+5% Tween 80+ddH2O at 3 mg/ml. But when water added, it turned yellowish green immediately. We are not sure if it was still fine in this vehicle. So we tried to use oil. 3mg of it dissolves in 40ul of DMSO clearly, and it can be dilute with corn oil at any proportion.
