nur für Forschungszwecke
LDC1267 Axl Inhibitor
Kat.-Nr.S7638
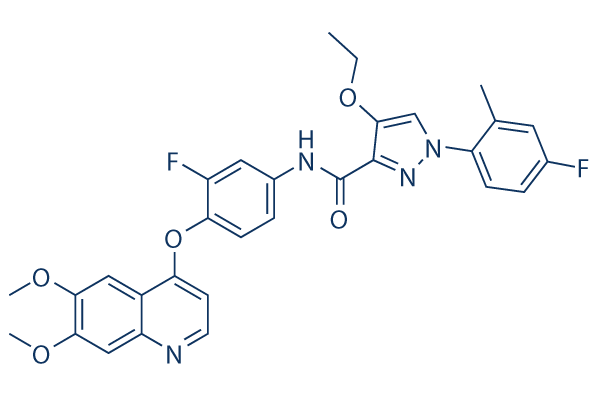
Chemische Struktur
Molekulargewicht: 560.55
Qualitätskontrolle
- In Nature Medicine für seine erstklassige Qualität zitiert
- COA
- NMR
- HPLC
- SDS
- Datenblatt
| Verwandte Ziele | EGFR VEGFR PDGFR FGFR c-Met Src MEK CSF-1R FLT3 HER2 |
|---|---|
| Weitere Axl Inhibitoren | Bemcentinib (R428) Dubermatinib(TP-0903) UNC2025 HCl CEP-40783 (RXDX-106) SGI-7079 Tamnorzatinib (ONO-7475) PF-07265807 RU-301 |
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 560.55 | Formel | C30H26F2N4O5 |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 1361030-48-9 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | N/A | Smiles | CCOC1=CN(N=C1C(=O)NC2=CC(=C(C=C2)OC3=C4C=C(C(=CC4=NC=C3)OC)OC)F)C5=C(C=C(C=C5)F)C | ||
Löslichkeit
|
In vitro |
DMSO
: 100 mg/mL
(178.39 mM)
Ethanol : 2 mg/mL Water : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Targets/IC50/Ki |
Mer
(Cell-free assay) <5 nM
Tyro3
(Cell-free assay) 8 nM
Axl
(Cell-free assay) 29 nM
|
|---|---|
| In vitro |
LDC1267 beeinflusst die Zellproliferation in 11 von 95 verschiedenen Zelllinien moderat mit einer IC50 von >5 μM. In NKG2D-aktivierten NK-Zellen hebt diese Verbindung die hemmenden Wirkungen der Gas6-Stimulation auf.
|
| Kinase-Assay |
Kinase-Bindungsassays
|
|
Zur Optimierung von Axl/TAM-Rezeptor-Inhibitoren wird ein Axl-Bindungsassay etabliert (HTRF-Methode; Kinase-Tracer 236). Dieser Assay basiert auf der Bindung und Verdrängung des Alexa Fluor 647-markierten Kinase-Tracers 236 an jede im Bindungsassay verwendete Glutathion-S-Transferase (GST)-markierte Kinase. Die Bindung des Tracers an die Kinase wurde unter Verwendung von Europium (Eu)-markierten Anti-GST-Antikörpern nachgewiesen. Die gleichzeitige Bindung sowohl des fluoreszierenden Tracers als auch der Eu-markierten Antikörper an die GST-markierte Kinase erzeugt ein Fluoreszenzresonanzenergietransfer (FRET)-Signal. Die Bindung des Inhibitors an die Kinase konkurriert mit der Bindung des Tracers, was zu einem Verlust des FRET-Signals führt. Für den Assay wird diese Verbindung in 20 mM HEPES, pH 8,0, 1 mM DTT, 10 mM MgCl2 und 0,01 % Brij35 verdünnt. Anschließend werden die interessierende Kinase (5 nM Endkonzentration), der fluoreszierende Tracer (15 nM Endkonzentration) und der LanthaScreen Eu-Anti-GST-Antikörper (2 nM Endkonzentration) mit den jeweiligen Verbindungsverdünnungen (von 5 nM bis 10 μM) gemischt und 1 Stunde lang inkubiert. Das FRET-Signal wird mit einem EnVision Multilabelreader 2104 quantifiziert.
|
|
| In vivo |
Bei B16F10-Melanom-tragenden Mäusen verbessert LDC1267 (20 mg/kg, i.p.) effizient die anti-metastatische NK-Zellaktivität und weist Tumormetastasen ohne ernsthafte Zytotoxizität zurück.
|
Literatur |
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
