nur für Forschungszwecke
BMH-21 DNA/RNA Synthesis Inhibitor
Kat.-Nr.S7718
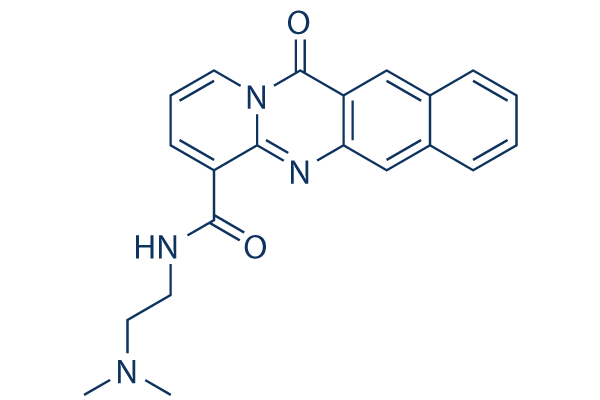
Chemische Struktur
Molekulargewicht: 360.41
Qualitätskontrolle
| Verwandte Ziele | HDAC PARP ATM/ATR DNA-PK WRN Topoisomerase PPAR Sirtuin Casein Kinase eIF |
|---|---|
| Weitere DNA/RNA Synthesis Inhibitoren | CX-5461 (Pidnarulex) B02 SCR7 Favipiravir (T-705) EED226 RK-33 Triapine (3-AP) Carmofur YK-4-279 Halofuginone |
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 360.41 | Formel | C21H20N4O2 |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 896705-16-1 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | N/A | Smiles | CN(C)CCNC(=O)C1=CC=CN2C1=NC3=CC4=CC=CC=C4C=C3C2=O | ||
Löslichkeit
|
In vitro |
DMSO
: 2 mg/mL
(5.54 mM)
Mit 50°C Wasserbad erwärmt;
Ultraschallbehandelt;
Ethanol : 2 mg/mL Water : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Targets/IC50/Ki |
RNA polymerase I
|
|---|---|
| In vitro |
BMH-21 verursacht den proteasomabhängigen Abbau von RPA194, dem großen katalytischen Untereinheitsprotein des Pol I-Holokomplexes. In der U2OS-Krebszelllinie führt diese Verbindung zur Degradation von RPA194 und zur Translokation von NCL mit einer IC50 von 0,05 μM bzw. 0,07 μM. Durch die Induktion von nukleolärem Stress zeigt sie auch eine starke Hemmung der Zellviabilität. |
| In vivo |
Diese Verbindung reduziert das Tumorwachstum in Maus-Xenografts. |
Literatur |
|
Anwendungen
| Methoden | Biomarker | Bilder | PMID |
|---|---|---|---|
| Western blot | MDM2 / p53 / p-p53 Bax / Caspase-3 / Cleaved caspase-3 |
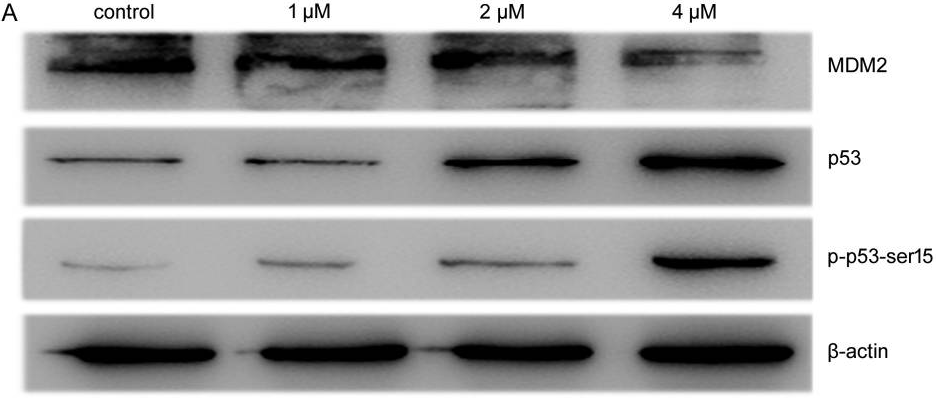
|
28656213 |
| Growth inhibition assay | Cell viability |
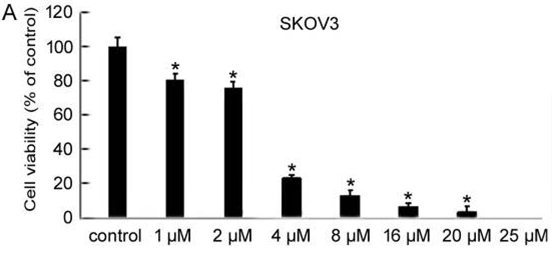
|
28656213 |
| Immunofluorescence | Nuclephosmin Fibrillarin |
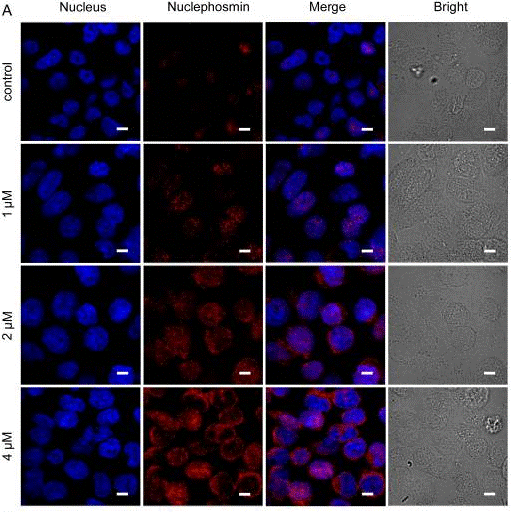
|
28656213 |
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
