nur für Forschungszwecke
Refametinib (RDEA119) MEK Inhibitor
Kat.-Nr.S1089
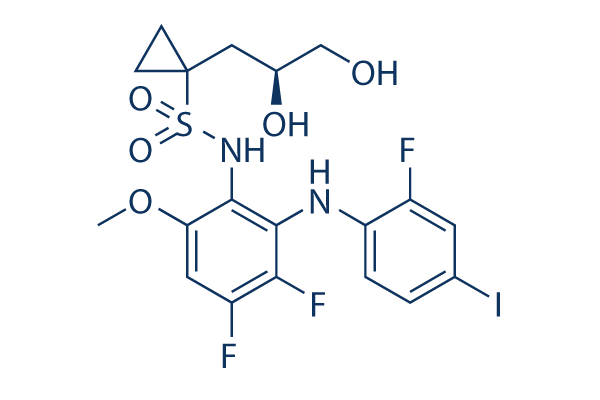
Chemische Struktur
Molekulargewicht: 572.34
Qualitätskontrolle
| Verwandte Ziele | ERK p38 MAPK Raf JNK Ras KRas S6 Kinase MAP4K TAK1 Mixed Lineage Kinase |
|---|---|
| Weitere MEK Inhibitoren | PD0325901 (Mirdametinib) U0126-EtOH PD 98059 PD184352 (CI-1040) BIX 02189 Pimasertib (AS-703026) TAK-733 AZD8330 SL-327 BIX 02188 |
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 572.34 | Formel | C19H20F3IN2O5S |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 923032-37-5 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | Bay 86-9766 | Smiles | COC1=CC(=C(C(=C1NS(=O)(=O)C2(CC2)CC(CO)O)NC3=C(C=C(C=C3)I)F)F)F | ||
Löslichkeit
|
In vitro |
DMSO
: 100 mg/mL
(174.72 mM)
Ethanol : 100 mg/mL Water : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Targets/IC50/Ki |
MEK1
(Cell-free assay) 19 nM
MEK2
(Cell-free assay) 47 nM
|
|---|---|
| In vitro |
Refametinib (RDEA119) bindet selektiv direkt an eine allosterische Tasche in den MEK1/2-Enzymen und ist hochwirksam bei der Hemmung der Zellproliferation in mehreren Tumorzelllinien, einschließlich A375, SK-MEI-28, Colo205, HT-29 und BxPC3. Es hemmt das Verankerungs-abhängige Wachstum menschlicher Krebszelllinien, die die Gain-of-Function-V600E-BRAF-Mutante beherbergen, mit GI50-Werten im Bereich von 67 bis 89 nM. Unter Verankerungs-unabhängigen Bedingungen sind die GI50-Werte für alle getesteten Zelllinien ähnlich (40-84 nM). Diese Verbindung zeigt eine Gewebeselektivität, die ihr Potenzial für Nebenwirkungen im Zusammenhang mit dem zentralen Nervensystem reduziert. Es hemmt potent die Proliferation der 4 Zelllinien, die eine BRAF-Mutation aufwiesen, hat aber keine oder geringe Auswirkungen auf die anderen 4 Zellen, die Wildtyp-BRAF aufwiesen (IC50 von 0,034-0,217 μM vs. 1,413-34,120 μM). Die hemmende Wirkung von Refametinib in ausgewählten Zelllinien OCUT1 (BRAF V600E(+), PIK3CA H1047R(+)) und SW1376 (BRAF V600E(+)) wird durch die Kombination mit dem mTOR-Inhibitor Temsirolimus verstärkt. Es und Temsirolimus zeigen auch synergistische Effekte auf den autophagischen Tod von OCUT1- und KAT18-Zellen, die selektiv getestet wurden.
|
| Kinase-Assay |
MEK-Kinase-Assay
|
|
Kinase-inaktive murine ERK2 (mERK2) K52A/T183A wird aus Escherichia coli, die mit dem pET21a-Vektor exprimiert wurden, affinitätsgereinigt. Die MEK1-Kinaseaktivität wird unter Verwendung von mERK2 K52A T183A als Substrat bestimmt. Rekombinantes MEK1-Enzym (5 nM) wird zunächst durch 0,02 Einheiten oder 1,5 nM RAF1 in Gegenwart von 25 mM HEPES (pH 7,8), 1 mM MgCl2, 50 mM NaCl, 0,2 mM EDTA und 50 μM ATP für 30 Minuten bei 25 °C aktiviert. Die Reaktionen werden durch Zugabe von 2 μM mERK2K52A T183A und 2,5 μCi [γ-33P] ATP in einem Gesamtvolumen von 20 μL gestartet. Die MEK2-Kinaseaktivität wird ähnlich bestimmt, außer dass die Aktivierung durch RAF1 nicht erforderlich ist und 11 nM MEK2-Enzym (aktiv) in den Assays verwendet werden. Das Kinase-Profiling für Refametinib (RDEA119) wird von Invitrogen unter Verwendung ihres Select Screen Kinase Profiling Service durchgeführt. Der Z'-LYTE-Biochemie-Assay wird verwendet. Diese Verbindung wird in Vierfachbestimmung bei 10 μM gegen 205 Kinasen getestet.
|
|
| In vivo |
Die orale Verabreichung von RDEA119 in einer Dosis von 50 mg/kg einmal täglich für 14 Tage führt zu einer 68%igen Tumorwachstumshemmung (TGI) im humanen Melanom-A375-Tumormodell. Diese Verbindung in einer Dosis von 25 mg/kg einmal täglich für 14 Tage führt zu einer 123%igen TGI im humanen Kolonkarzinom-Colo205-Tumormodell (TGI > 100% tritt auf, wenn der Tumor unter sein Ausgangsvolumen schrumpft). Eine Dosis von 25 mg/kg einmal täglich für 14 Tage erzeugt 56% und 67% TGI für HT-29- bzw. A431-Tumoren.
|
Literatur |
|
Anwendungen
| Methoden | Biomarker | Bilder | PMID |
|---|---|---|---|
| Growth inhibition assay | IC50 |
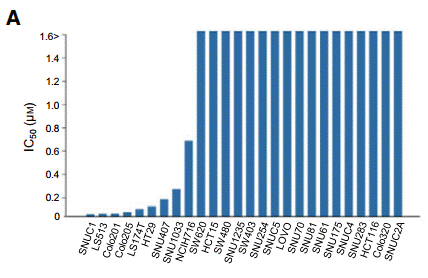
|
29896883 |
| Western blot | p-BRAF / BRAF / p-MEK / MEK / p-ERK / ERK Cyclin D1 / Cyclin E / p27 p-STAT3 / STAT3 |
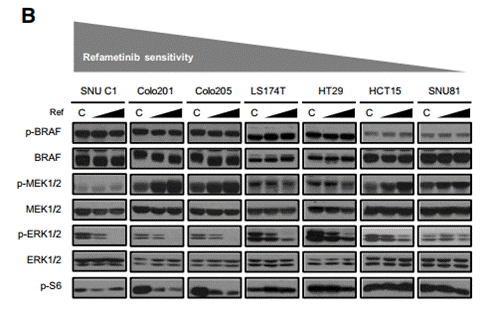
|
29896883 |
Klinische Studieninformationen
(Daten von https://clinicaltrials.gov, aktualisiert am 2024-05-22)
| NCT-Nummer | Rekrutierung | Erkrankungen | Sponsor/Kooperationspartner | Startdatum | Phasen |
|---|---|---|---|---|---|
| NCT01392521 | Completed | Neoplasms |
Bayer |
July 2011 | Phase 1 |
| NCT00785226 | Completed | Advanced Cancer |
Bayer |
November 2008 | Phase 1|Phase 2 |
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
