nur für Forschungszwecke
RG108 DNA Methyltransferase Inhibitor
Kat.-Nr.S2821
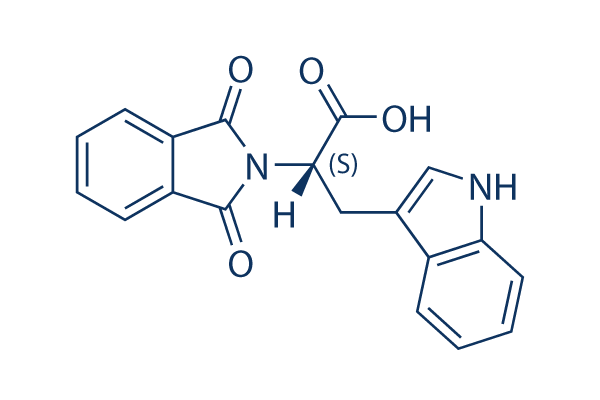
Chemische Struktur
Molekulargewicht: 334.33
Qualitätskontrolle
| Verwandte Ziele | HDAC JAK BET Histone Methyltransferase PKC PARP HIF PRMT EZH2 AMPK |
|---|---|
| Weitere DNA Methyltransferase Inhibitoren | SGI-1027 Zebularine (NSC 309132) GSK3685032 Gamma-Oryzanol CM272 β-thujaplicin Bobcat339 DC-05 2'-Deoxy-5-Fluorocytidine GSK-3484862 |
Zellkultur, Behandlung & Arbeitskonzentration
| Zelllinien | Assay-Typ | Konzentration | Inkubationszeit | Formulierung | Aktivitätsbeschreibung | PMID |
|---|---|---|---|---|---|---|
| human THP1 cells | Function assay | Inhibition of ACAT-mediated esterified cholesterol accumulation in human THP1 cells exposed to acetyl-LDL during differentiation assessed as effect on foam cell formation, IC50=1.5 μM | 18620381 | |||
| human HepG2 cells | Function assay | Inhibition of ACAT in human HepG2 cells, IC50=0.479 μM | 19167888 | |||
| rat macrophages | Function assay | 24 h | Inhibition of ACAT in rat macrophages assessed as incorporation of extracellular [3H]-oleic acid-BSA complex into the intracellular cholesteryl ester after 24 hrs, IC50=0.479 μM | 19464189 | ||
| MCF7 | Apoptosis assay | 10 uM | 6 hrs | Induction of apoptosis in human MCF7 cells assessed as apoptotic cells at 10 uM after 6 hrs by FITC-Annexin V/propidium iodide staining-based FACS analysis relative to control | 25801160 | |
| MCF7 | Apoptosis assay | 10 uM | 6 hrs | Induction of apoptosis in human MCF7 cells assessed as dead cells at 10 uM after 6 hrs by FITC-Annexin V/propidium iodide staining-based FACS analysis relative to control | 25801160 | |
| MCF7 | Function assay | 100 uM | 1 hr | Competitive inhibition of DNMT1 in (S)-Methyl 2-(2,5-dioxo-2,5-dihydro-1H-pyrrol-1-yl)-3-(5-(prop-2-yn-1-yloxy)-1H-indol-3-yl)propanoate labeled human MCF7 cells at 100 uM preincubated for 1 hr followed by cell labeling for 30 mins and clicked with TER-PE | 25801160 | |
| MCF7 | Function assay | 50 to 500 uM | 1 hr | Competitive inhibition of DNMT1 in (S)-Methyl 2-(2,5-dioxo-2,5-dihydro-1H-pyrrol-1-yl)-3-(5-(prop-2-yn-1-yloxy)-1H-indol-3-yl)propanoate labeled human MCF7 cells at 50 to 500 uM preincubated for 1 hr followed by cell labeling for 1 hr and clicked with TER | 25801160 | |
| SK-N-MC | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for SK-N-MC cells | 29435139 | |||
| Klicken Sie hier, um weitere experimentelle Daten zu Zelllinien anzuzeigen | ||||||
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 334.33 | Formel | C19H14N2O4 |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 48208-26-0 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | N-Phthalyl-L-tryptophan | Smiles | C1=CC=C2C(=C1)C(=O)N(C2=O)C(CC3=CNC4=CC=CC=C43)C(=O)O | ||
Löslichkeit
|
In vitro |
DMSO
: 66 mg/mL
(197.4 mM)
Ethanol : 66 mg/mL Water : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Targets/IC50/Ki |
DNA methyltransferase
(Cell-free assay) 115 nM
|
|---|---|
| In vitro |
RG108 blockiert effektiv DNA methyltransferases in vitro und verursacht keine kovalente Enzymbindung in menschlichen Zelllinien. Die Inkubation von Zellen mit niedrigen mikromolaren Konzentrationen dieser Verbindung führt zu einer signifikanten Demethylierung der genomischen DNA ohne nachweisbare Toxizität. Faszinierenderweise führt es zur Demethylierung und Reaktivierung von Tumorsuppressorgenen, beeinflusst jedoch nicht die Methylierung zentromerer Satellitensequenzen. In einer anderen Studie wird die Synthese und In-vitro-Analyse eines biotinylierten Konjugats dieser Chemikalie untersucht, um die Wechselwirkungen mit DNA methyltransferase-Enzymen zu bewerten. In einer kürzlich durchgeführten Studie wurde gezeigt, dass diese Verbindung die DNA methyltransferases-Aktivität in SM-abgeleiteten iPS-Zellen im Vergleich zu nativen SMs signifikant reduzieren kann.
|
| Kinase-Assay |
In-vitro-Methylierungsassay
|
|
Die Substrat-DNA für den In-vitro-Methylierungsassay ist ein 798 bp Fragment (−423/+375 relativ zum Initiationskodon) aus der Promotorregion des menschlichen p16Ink4a-Gens. Die Methylierungsreaktion enthält 350 bis 400 ng Substrat-DNA und 4 Einheiten M.SssI-Methylase (0,5 μM) in einem Endvolumen von 50 μL. Diese Verbindung wird zu Endkonzentrationen von 10, 100, 200 und 500 μM hinzugefügt. Die Reaktionen werden bei 37 °C für 2 Stunden durchgeführt. Nach Abschluss wird die Reaktion bei 65 °C für 15 Minuten inaktiviert und die DNA mit einem PCR-Reinigungskit gereinigt. Dreihundert Nanogramm gereinigter DNA werden 3 Stunden lang bei 60 °C mit 30 Einheiten BstUI verdaut und auf 2% Tris-Borat-EDTA-Agarosegelen analysiert.
|
Literatur |
|
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
