nur für Forschungszwecke
Chaetocin Histone Methyltransferase Inhibitor
Kat.-Nr.S8068
Chemische Struktur
Molekulargewicht: 696.84
Qualitätskontrolle
| Verwandte Ziele | HDAC JAK BET PKC PARP HIF PRMT EZH2 AMPK Histone Acetyltransferase |
|---|---|
| Weitere Histone Methyltransferase Inhibitoren | Pinometostat (EPZ5676) 3-Deazaneplanocin A (DZNep) Hydrochloride BIX-01294 trihydrochloride UNC1999 EPZ015666 (GSK3235025) EPZ004777 MM-102 (HMTase Inhibitor IX) SGC 0946 EPZ005687 UNC0638 |
Zellkultur, Behandlung & Arbeitskonzentration
| Zelllinien | Assay-Typ | Konzentration | Inkubationszeit | Formulierung | Aktivitätsbeschreibung | PMID |
|---|---|---|---|---|---|---|
| A549 | Cytotoxicity assay | Cytotoxicity against human A549 cells, IC50 = 0.025 μM. | 20303767 | |||
| Hep3b | Function assay | Inhibition of HIF-1alpha-mediated VEGF expression in human Hep3b cells by luciferase reporter gene assay, IC50 = 0.04 μM. | 22305612 | |||
| Hep3b | Function assay | Inhibition of HIF-1alpha-mediated VEGF secretion in human Hep3b cells by ELISA, IC50 = 0.1 μM. | 22305612 | |||
| Jurkat | Cytotoxicity assay | 40 hrs | Cytotoxicity against human Jurkat cells after 40 hrs bioluminescence assay, IC50 = 0.6 μM. | 20303767 | ||
| Drosophila melanogaster SL2 | Function assay | 0.1 uM | 5 days | Inhibition of SU(VAR)3-9 in Drosophila melanogaster SL2 cells assessed as reduction of H3K9me2 at 0.1 uM after 5 days by MALDI-TOF MS seeded at 2.5 million cells/ml density | 16408017 | |
| Drosophila melanogaster SL2 | Function assay | 0.5 uM | 5 days | Inhibition of SU(VAR)3-9 in Drosophila melanogaster SL2 cells assessed as reduction of H3K9me2 at 0.5 uM after 5 days by MALDI-TOF MS seeded at 2.5 million cells/ml density | 16408017 | |
| Drosophila melanogaster SL2 | Function assay | 0.5 uM | Inhibition of SU(VAR)3-9 in Drosophila melanogaster SL2 cells assessed as reduction of H3K9me3 at 0.5 uM by immunostaining method | 16408017 | ||
| HL60 | Apoptosis assay | 30 uM | 4 hrs | Induction of apoptosis in human HL60 cells assessed as necrotic cell death at 30 uM after 4 hrs using DAPI/PI by confocal laser microscopy analysis | 20675131 | |
| HL60 | Apoptosis assay | 0.3 uM | 4 hrs | Induction of apoptosis in human HL60 cells assessed as nuclear fragmentation at 0.3 uM after 4 hrs using DAPI/PI by confocal laser microscopy analysis | 20675131 | |
| HL60 | Apoptosis assay | 0.3 uM | 2 hrs | Induction of apoptosis in human HL60 cells assessed as activation of caspase 3 at 0.3 uM after 2 hrs by Western blot analysis | 20675131 | |
| MCF7 | Function assay | Inhibition of histone-lysine N-methyltransferase in human MCF7 cells assessed as decrease in methylation of RARbeta DNA by qPCR method | ChEMBL | |||
| Klicken Sie hier, um weitere experimentelle Daten zu Zelllinien anzuzeigen | ||||||
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 696.84 | Formel | C30H28N6O6S4 |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 28097-03-2 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
Löslichkeit
|
In vitro |
DMSO
: 25 mg/mL
(35.87 mM)
Water : Insoluble Ethanol : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Targets/IC50/Ki |
dSU(VAR)3-9
0.8 μM
mouse G9a
2.5 μM
Neurospora crassa DIM5
3 μM
|
|---|---|
| In vitro |
In SL-2 Drosophila-Gewebekulturzellen bewirkt Chaetocin die Hemmung von SU(VAR)3-9 und der Anzahl der an Lys9 dimethylierten H3-Moleküle und hemmt auch das Zellwachstum. In menschlichen HepG2-, Hep3B- und Huh7-Hepatomzellen hemmt diese Verbindung HIF-1-vermittelte hypoxische Reaktionen. Es hemmt auch potent die Proliferation und Koloniebildung in einer Vielzahl von Krebszelllinien mit IC50 von 2-10 nM. |
| Kinase-Assay |
Methylierungsassays
|
|
Sofern nicht anders angegeben, werden die Reaktionen wie folgt durchgeführt: 1 g gereinigtes Enzym (dSU(VAR)3-9 213; GST-hSUV39H1(82-412); GST-ncDIM5(19-318) und GSTmG9a(621-1000); dPRSET7(1-691); His-SET7/9(109-366) oder im Baculovirus-Expressionssystem exprimierter Enzymkomplex (dE(z), dSU(Z)12, dp55, dESC)) wurden 30 Minuten lang mit 1 g eines Peptids, das die ersten 19 Aminosäuren von H3 plus einen zusätzlichen Cysteinrest (ARTKQTARKSTGGKAPRKQC) enthält, in einem Gesamtvolumen von 40 μl in BC25 (10 mM HEPES pH 7,6, 25 mM NaCl, 1 mM EDTA, 10 % Glycerin) inkubiert. Alle Enzyme außer dem E(z)-Komplex hatten eine ähnliche spezifische Aktivität zwischen 5-10 nmol/min/mg. Für den E(z)-Komplex ist die spezifische Aktivität etwa 5-10-mal niedriger, was wahrscheinlich auf eine unvollständige Bildung des vollständigen Komplexes zurückzuführen ist, der für die höchste Aktivität erforderlich ist. Als Methyldonor ist S-Adenosyl[methyl-3H]methionin (SAM) in der Reaktion in einer Endkonzentration von 40 M mit einer spezifischen Aktivität von 0,3 Ci/mmol vorhanden. Die Reaktionen werden durch Zugabe von 1/10 Volumen 100%iger Essigsäure gestoppt und der Einbau von Radioaktivität wird durch Auftragen von 30 μl der Reaktion auf P81-Filterpapier und anschließendes Szintillationszählen gemessen. Für das Inhibitorscreening werden 1 μl dieser Verbindung mit einer Konzentration von 10 g/l zu jeder Reaktion gegeben. In den Fällen, in denen PRSET7 oder der E(z)-Komplex als Enzym verwendet wird, ersetzten rekombinante Nukleosomen (1 g) bzw. rekombinantes H3 (1 g) das H3-Peptid als Substrat.
|
|
| In vivo |
Bei Hepa 1c1c-7-Tumor tragenden Mäusen hemmt Chaetocin (0,25 mg/kg, i.p.) das Tumorwachstum durch Deregulierung der HIF-1[alpha]-vermittelten Angiogenese. Bei SKOV3-Tumor tragenden Nacktmäusen verzögert diese Verbindung (0,25 mg/kg, i.p.) das Tumorwachstum signifikant mit minimalen Anzeichen von Toxizität. |
Literatur |
|
Anwendungen
| Methoden | Biomarker | Bilder | PMID |
|---|---|---|---|
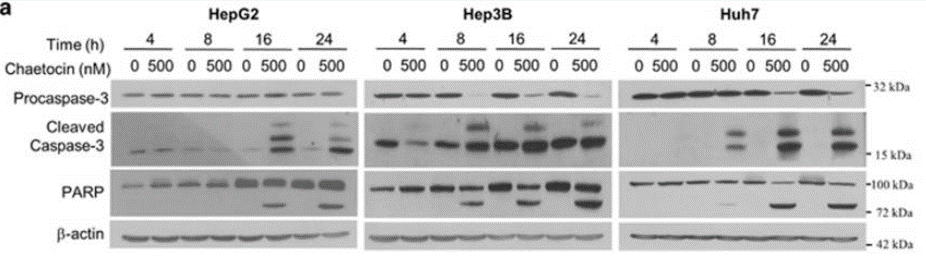
| Western blot | Procaspase-3 / Cleaved Caspase-3 / PARP |
|
26890137 |
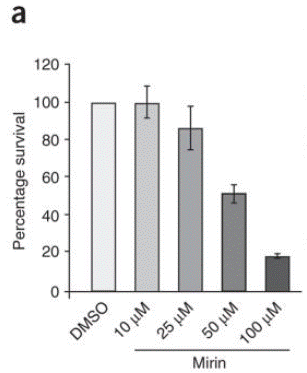
| Growth inhibition assay | Cell viability |
|
18176557 |
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
