nur für Forschungszwecke
UNC1999 Histone Methyltransferase Inhibitor
Kat.-Nr.S7165
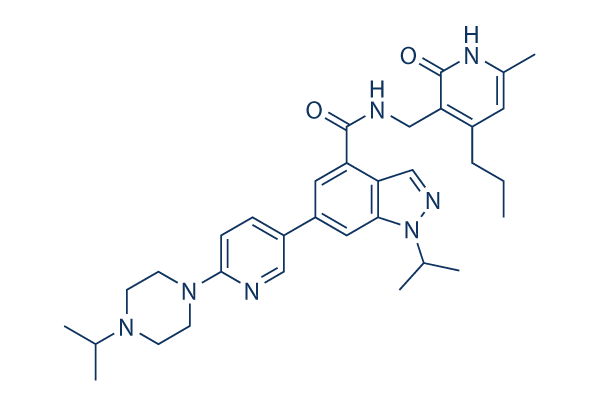
Chemische Struktur
Molekulargewicht: 569.74
Qualitätskontrolle
| Verwandte Ziele | HDAC JAK BET PKC PARP HIF PRMT EZH2 AMPK Histone Acetyltransferase |
|---|---|
| Weitere Histone Methyltransferase Inhibitoren | Pinometostat (EPZ5676) 3-Deazaneplanocin A (DZNep) Hydrochloride BIX-01294 trihydrochloride EPZ015666 (GSK3235025) EPZ004777 MM-102 (HMTase Inhibitor IX) Chaetocin SGC 0946 EPZ005687 UNC0638 |
Zellkultur, Behandlung & Arbeitskonzentration
| Zelllinien | Assay-Typ | Konzentration | Inkubationszeit | Formulierung | Aktivitätsbeschreibung | PMID |
|---|---|---|---|---|---|---|
| human MCF10A cells | Function assay | 72 h | Inhibition of EZH2 in human MCF10A cells assessed as reduction of H3K27me3 level after 72 hrs by Western blot analysis, IC50=0.124 μM | 25406853 | ||
| human MCF10A cells | Cytotoxic assay | Cytotoxicity against human MCF10A cells assessed as cell viability by Alamar Blue assay, EC50=19.2 μM | 25406853 | |||
| Klicken Sie hier, um weitere experimentelle Daten zu Zelllinien anzuzeigen | ||||||
Chemische Informationen, Lagerung & Stabilität
| Molekulargewicht | 569.74 | Formel | C33H43N7O2 |
Lagerung (Ab dem Eingangsdatum) | |
|---|---|---|---|---|---|
| CAS-Nr. | 1431612-23-5 | SDF herunterladen | Lagerung von Stammlösungen |
|
|
| Synonyme | N/A | Smiles | CCCC1=C(C(=O)NC(=C1)C)CNC(=O)C2=C3C=NN(C3=CC(=C2)C4=CN=C(C=C4)N5CCN(CC5)C(C)C)C(C)C | ||
Löslichkeit
|
In vitro |
DMSO
: 100 mg/mL
(175.51 mM)
Ethanol : 100 mg/mL Water : Insoluble |
Molaritätsrechner
|
In vivo |
|||||
In-vivo-Formulierungsrechner (Klare Lösung)
Schritt 1: Geben Sie die untenstehenden Informationen ein (Empfohlen: Ein zusätzliches Tier zur Berücksichtigung von Verlusten während des Experiments)
Schritt 2: Geben Sie die In-vivo-Formulierung ein (Dies ist nur der Rechner, keine Formulierung. Bitte kontaktieren Sie uns zuerst, wenn es im Abschnitt "Löslichkeit" keine In-vivo-Formulierung gibt.)
Berechnungsergebnisse:
Arbeitskonzentration: mg/ml;
Methode zur Herstellung der DMSO-Stammlösung: mg Wirkstoff vorgelöst in μL DMSO ( Konzentration der Stammlösung mg/mL, Bitte kontaktieren Sie uns zuerst, wenn die Konzentration die DMSO-Löslichkeit der Wirkstoffcharge überschreitet. )
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügenμL PEG300, mischen und klären, dann hinzufügenμL Tween 80, mischen und klären, dann hinzufügen μL ddH2O, mischen und klären.
Methode zur Herstellung der In-vivo-Formulierung: Nehmen Sie μL DMSO Stammlösung, dann hinzufügen μL Maisöl, mischen und klären.
Hinweis: 1. Bitte stellen Sie sicher, dass die Flüssigkeit klar ist, bevor Sie das nächste Lösungsmittel hinzufügen.
2. Achten Sie darauf, das/die Lösungsmittel der Reihe nach hinzuzufügen. Sie müssen sicherstellen, dass die bei der vorherigen Zugabe erhaltene Lösung eine klare Lösung ist, bevor Sie mit der Zugabe des nächsten Lösungsmittels fortfahren. Physikalische Methoden wie Vortex, Ultraschall oder ein heißes Wasserbad können zur Unterstützung des Lösens verwendet werden.
Wirkmechanismus
| Merkmale |
The first orally bioavailable inhibitor against wild-type and mutant EZH2 as well as EZH1.
|
|---|---|
| Targets/IC50/Ki |
EZH2
(Cell-free assay) 2 nM
EZH1
(Cell-free assay) 45 nM
|
| In vitro |
UNC1999 ist in vitro hochwirksam gegen EZH2 Y641N- und EZH2 Y641F-Mutanten. Diese Verbindung verursacht konzentrationsabhängige Reduktionen von H3K27me3 in MCF10A-Zellen mit einer IC50 von 124 nM, während sie eine geringe zelluläre Toxizität aufweist. Sie zeigt eine potente, konzentrationsabhängige Hemmung der Zellproliferation mit einer EC50 von 633 nM in einer DLBCL-Zelllinie, die die EZH2Y641N-Mutante beherbergt. Darüber hinaus reichert biotinyliertes UNC1999 EZH2 aus HEK293T-Zelllysaten an und kann somit in chemoproteomischen Studien verwendet werden.
|
| Kinase-Assay |
Szintillations-Proximity-Assay
|
|
Methyltransferase-Aktivitätsassays werden durch Überwachung des Einbaus einer tritiummarkierten Methylgruppe aus S-Adenosylmethionin (3H-SAM) in biotinylierte Peptidsubstrate unter Verwendung des Scintillation Proximity Assay (SPA) für den PRC2-EZH2-Trimerkomplex (EZH2:EED:SUZ12), den PRC2-EZH1-Pentamerkomplex (EZH1:EED:SUZ12:RBBP4:AEBP2), SETD7, G9a, GLP, SETDB1, SETD8, SUV420H1, SUV420H2, SUV39H2, den MLL1-Tetramerkomplex (MLL:WDR5:RbBP5:ASH2L), PRMT1, PRMT3, PRMT5-MEP50-Komplex und SMYD2 durchgeführt. Der Reaktionspuffer für SMYD2 und SMYD3 ist 50 mM Tris pH 9,0, 5 mM DTT, 0,01 % TritonX-100; für G9a, GLP und SUV39H2 ist 25 mM Kaliumphosphat pH 8,0, 1 mM EDTA, 2 mM MgCl2 und 0,01 % Triton X-100; und für andere HMTs 20 mM Tris pH 8,0, 5 mM DTT, 0,01 % TritonX-100. Um die enzymatischen Reaktionen zu stoppen, werden 10 μL 7,5 M Guanidinhydrochlorid hinzugefügt, gefolgt von 180 μL Puffer, gemischt und auf eine 384-Well-FlashPlate übertragen. Nach dem Mischen werden die Reaktionsgemische inkubiert und die CPM-Werte mit einem Topcount-Plattenlesegerät gemessen. Die CPM-Werte ohne diese Verbindung für jeden Datensatz werden als 100 % Aktivität definiert. Ohne das Enzym werden die CPM-Werte in jedem Datensatz als Hintergrund (0 %) definiert. IC50-Werte werden unter Verwendung von Verbindungskonzentrationen im Bereich von 100 nM bis 100 μM bestimmt. Die IC50-Werte werden mit der SigmaPlot-Software bestimmt. EZH2-Y641F-Assays werden unter Verwendung von 30 nM Enzym in 20 mM Tris pH 8, 5 mM DTT, 0,01 % Triton X-100, 5 μM SAM und 1 μM H3 (1-24)-Peptid (wie für den Wildtyp-PRC2-EZH2-Komplex) durchgeführt. Für DNMT1 wird der Assay unter Verwendung von hemimethylierter dsDNA als Substrat durchgeführt. Das dsDNA-Substrat wird durch Annealing zweier komplementärer Stränge (biotinylierter Vorwärtsstrang: BGAGCCCGTAAGCCCGTTCAGGTCG und Reverse-Strang: CGACCTGAACGGGCTTACGGGCTC), synthetisiert von Eurofins MWG Operon, hergestellt. Der Reaktionspuffer ist 20 mM Tris-HCl, pH 8,0, 5 mM DTT, 0,01 % Triton X-100. Methyltransferase-Aktivitätsassays für DOT1L werden unter Verwendung von Filterplatten durchgeführt. Reaktionsgemische in 20 mM Tris-HCl, pH 8,0, 5 mM DTT, 2 mM MgCl2 und 0,01 % Triton X-100 werden 1 Stunde bei Raumtemperatur inkubiert, 100 μL 10 % TCA werden hinzugefügt, gemischt und auf eine Filterplatte übertragen. Die Platten werden 2 Minuten bei 2000 U/min zentrifugiert, gefolgt von 2 weiteren 10 % TCA-Wäschen und einer Ethanolwäsche (180 μL) gefolgt von Zentrifugation. Die Platten werden getrocknet und 100 μL MicroO werden hinzugefügt und zentrifugiert. 70 μL MicroO werden hinzugefügt und die CPM-Werte werden mit einem Topcount-Plattenlesegerät gemessen.
|
|
| In vivo |
Die Behandlung mit UNC1999 (150 und 50 mg/kg, i.p.) führt zu Plasmakonzentrationen dieser Verbindung über ihrer zellulären IC50 über 24 Stunden in vivo. Darüber hinaus ist sie bei Mäusen auch oral bioverfügbar, was chronische Tierstudien praktischer und bequemer macht.
|
Literatur |
|
Anwendungen
| Methoden | Biomarker | Bilder | PMID |
|---|---|---|---|
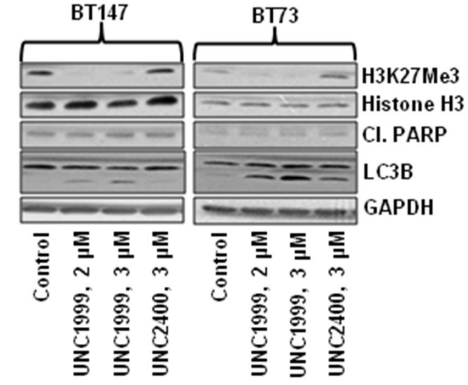
| Western blot | H3K27Me3 / Cleaved PARP / LC3B H3K27me2 / H3K27me1 / H3K27ac / H3K4me3 / H3K9me2 / H3K36me2 |
|
27449082 |
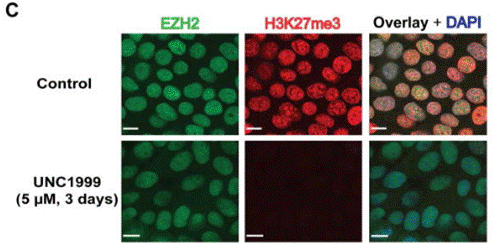
| Immunofluorescence | EZH2 / H3K27me3 NF-κB |
|
23614352 |
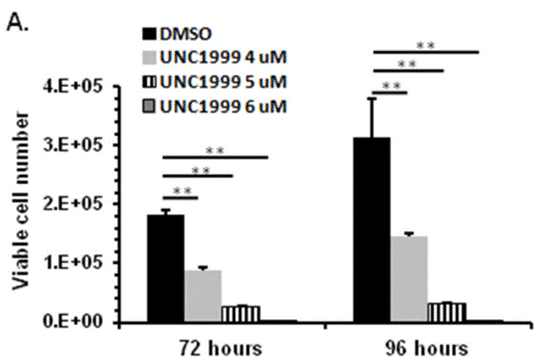
| Growth inhibition assay | Cell viability |
|
27449082 |
Technischer Support
Tel: +1-832-582-8158 Ext:3
Wenn Sie weitere Fragen haben, hinterlassen Sie bitte eine Nachricht.
